Генетическому алфавиту добавили две буквы
С двумя буквами подопытные бактерии получили и два новых генетических слова.
Фотографий со светящимися бактериями на свете хоть пруд пруди, но именно эти засветились потому, что сумели синтезировать белок, закодированный с использованием совершенно новых генетических «слов». (Фото: Bill Kiosses / The Scripps Research Institute.)
Азотистые основания – генетические «буквы» – спариваются в цепях ДНК по определенным правилам, тем самым поддерживая постоянную структуру всей молекулы. (Иллюстрация: Richard Wheeler / Wikipedia.)
‹
›
Открыть в полном размере
У всех живых организмов, от бактерий до человека, генетический алфавит состоит из четырех молекулярных букв – А, Т, Г, Ц. Буквы эти – азотистые основания аденин, тимин, гуанин и цитозин. В двуцепочечной молекуле ДНК они пришиты к длиннейшим сахарофософатным «перилам» и смотрят друг на друга – то есть в пространство между цепочек, причем напротив А всегда должен стоять Т, а напротив Г – Ц.
Но сами по себе буквы ничего не значат – они должны складываться в слова. Слова в генетическом коде земной жизни состоят из трех букв, образующих между собой разные комбинации. Такие слова-триплеты соответствуют аминокислотам, и когда идет синтез белка, то специальные машины читают код тройками, и в соответствии с последовательностью троек строят последовательность аминокислот. (Там есть особенности с передачей информации от ДНК через матричную РНК к белоксинтезирующим машинам, но эти подробности мы сейчас опустим.)
Если посчитать, сколько комбинаций-триплетов можно получить, имея на руках четыре буквы, то мы получим 64 комбинации. Между тем в синтезе белков участвуют всего 20 аминокислот. То есть код сильно избыточен, и потому одной аминокислоте часто соответствуют два, три и больше генетических слов.
Рано или поздно биологам должны была прийти в голову мысль, что генетический код можно отредактировать на фундаментальном уровне, то есть приписать каким-то словам новые значения, а то и вовсе расширить алфавит и словарь.
Тем не менее, несколько лет назад в Science вышла статья, в которой описывалось, как у бактерий кишечной палочки удалось присвоить одному из избыточных триплетов значение абсолютно новой аминокислоты. Поскольку аминокислоты к белоксинтезирующей машине подвозят специальные транспортные РНК (тРНК), то пришлось создать и новую тРНК, которая носила бы с собой новую аминокислоту и распознавала бы ее код во время синтеза полипептидной цепи.
Но тРНК не сами хватают свои аминокислоты, для этого есть специальные ферменты – аминоацил-тРНК-синтетазы, которые узнают только свои аминокислоты и соответствующие им тРНК. Понятно, что авторам работы пришлось создать тут и новый фермент, который бы соединял новую аминокислоту с новой тРНК.
Но можно, как мы сказали, начать редактировать код еще раньше – с алфавита. Иными словами, что мы сначала должны создать новую букву, то есть новое азотистое основание. На самом деле, с химической точки зрения тут особо сложного ничего нет, и создать новую букву можно, просто модифицировав какую-нибудь старую. (Более того, клетки сами модифицируют «буквы», и порой довольно сильно, правда, код от этого не меняется.)
Иными словами, что мы сначала должны создать новую букву, то есть новое азотистое основание. На самом деле, с химической точки зрения тут особо сложного ничего нет, и создать новую букву можно, просто модифицировав какую-нибудь старую. (Более того, клетки сами модифицируют «буквы», и порой довольно сильно, правда, код от этого не меняется.)
Но мало синтезировать новую «букву», она должна нормально встроиться в нуклеиновую кислоту и участвовать во всех необходимых молекулярных процессах. Впервые такие «буквы» появились в конце 80-х годов прошлого века, то были модификации гуанина и цитозина; ДНК с ними нормально удваивалась и на ней нормально синтезировалась молекула-посредник – матричная РНК, которая несет информацию о белке к белоксинтезирующему аппарату.
Флойд Ромесберг (Floyd Romesberg) и его коллеги из Института Скриппса за последние двадцать лет создали несколько сотен новых букв, однако расширить с их помощью генетический код удалось только сейчас. В своей последней работе исследователи вместе с сотрудниками биотехнологической компании Synthorx использовали два новых азотистых основания, которые для простоты лучше назвать X и Y.
В своей последней работе исследователи вместе с сотрудниками биотехнологической компании Synthorx использовали два новых азотистых основания, которые для простоты лучше назвать X и Y.
В двуцепочечной молекуле ДНК они стоят друг напротив друга, но при этом соединяются они не так, как все остальные: если А и Т, Г и Ц держатся друг за друга водородными связями, то X и Y удерживаются друг напротив друга гидрофобными силами, что добавляет им странности. Тем не менее, пара X и Y не выбивается из ряда прочих «традиционных» пар, а значит, не нарушает структуру ДНК – ее физические параметры (расстояния между цепями, размер шага спирали и т. д.) остаются по всей длине прежними, что очень важно для взаимодействия с белками и вообще для целостности молекулы.
Поначалу, впрочем, бактерии с такой ДНК делились медленнее обычного и норовили заменить «нетрадиционную» ДНК нормальной. Однако исследователи все-таки доработали новые буквы, так что они перестали доставлять неудобство бактериям. Теперь осталось только сделать из них новые слова-триплеты, которые кодировали бы две аминокислоты. В качестве новых аминокислот выбрали модификации лизина и фенилаланина. Новые триплеты генетических букв оказались такими: AXC (то есть аденин-X-цитозин) и GYT (гуанин-Y-тимин). Их внедрили в ДНК, кодирующую зеленый флуоресцентный белок, а ДНК отправили в бактериальную клетку – и клетка засветилась зеленым. При этом, разумеется, у бактерий были вышеупомянутые ферменты и тРНК, чтобы обслуживать новые триплеты в коде и новые аминокислоты, а сами аминокислоты нужно было добавлять в питательную среду. Полностью результаты описаны в статье в Nature.
Теперь осталось только сделать из них новые слова-триплеты, которые кодировали бы две аминокислоты. В качестве новых аминокислот выбрали модификации лизина и фенилаланина. Новые триплеты генетических букв оказались такими: AXC (то есть аденин-X-цитозин) и GYT (гуанин-Y-тимин). Их внедрили в ДНК, кодирующую зеленый флуоресцентный белок, а ДНК отправили в бактериальную клетку – и клетка засветилась зеленым. При этом, разумеется, у бактерий были вышеупомянутые ферменты и тРНК, чтобы обслуживать новые триплеты в коде и новые аминокислоты, а сами аминокислоты нужно было добавлять в питательную среду. Полностью результаты описаны в статье в Nature.
Возникает вопрос, зачем вообще нужно столь фундаментально вмешиваться в генетический алфавит и словарь. Здесь все просто: с новыми аминокислотами можно создавать новые белковые молекулы, сконструированные для конкретных задач. Конечно, белки модифицировали в лабораториях и раньше, но, внедрившись в генетический код, это можно проделывать намного эффективнее. И тут речь не только о белках, но и о полусинтетических организмах с новыми свойствами.
И тут речь не только о белках, но и о полусинтетических организмах с новыми свойствами.
Причем не стоит забывать, что, добавив всего лишь две буквы, мы расширяем число возможных генетических слов до 216, и в результате получаем возможность кодировать еще 172 аминокислоты, вдобавок к прежним двадцати. Простор для биоинженерии, как видим, бескрайний.
Генетический алфавит расширен. Или нет?
Дмитрий Жарков
«Наука из первых рук» №1(61), 2015
Об автореДмитрий Олегович Жарков — доктор биологических наук, заведующий группой взаимодействий биополимеров Института химической биологии и фундаментальной медицины СО РАН (Новосибирск). В течение восьми лет работал в лаборатории А. Гроллмана (США). Автор и соавтор 80 научных работ. |
В десятку наиболее выдающихся результатов 2014 г. вошли работы исследователей из Скриппсовского института (Ла-Холья, США), которые не только создали ДНК с двумя новыми «буквами» генетического кода, но и заставили ее воспроизводиться внутри кишечной палочки — традиционного экспериментального объекта молекулярных биологов.
Как всем известно со школьной скамьи, «текст» молекулы ДНК — хранилища наследственной информации — написан всего четырьмя «буквами»: A, T, G и C. Буквы эти обозначают четыре азотистых основания — аденин, тимин, гуанин и цитозин, которые крепятся к сахарофосфатному остову. Две такие конструкции и составляют знаменитую двойную спираль ДНК, которая в итоге содержит все инструкции по постройке и работе нашего организма.
Две цепи ДНК связаны друг с другом по принципу комплементарности: напротив A в одной цепи всегда стоит T в другой, а напротив G — всегда C. Между «противоположными» основаниями в этих парах образуются так называемые водородные связи, что и определяет соответствие оснований друг другу. Например, между A и C такие связи образоваться не могут, поэтому и говорят, что основания подходят друг к другу как ключ к замку.
Структура ДНК стала известна еще в середине прошлого века, и уже тогда ученые стали задаваться вопросами: почему живая природа использует именно эти две пары оснований, а не какие-нибудь другие, и нельзя ли как-нибудь «подкрутить винтики» в клетке, чтобы заставить ее использовать другие основания? Ответ на первый вопрос не получен до сих пор. Многие специалисты в области предбиологической эволюции предполагают, что такой выбор был случайностью, которая закрепилась в дальнейшем, когда был пройден самый трудный этап возникновения жизни на нашей планете — когда молекулы стали «репликаторами», т. е. научились воспроизводить сами себя.
Многие специалисты в области предбиологической эволюции предполагают, что такой выбор был случайностью, которая закрепилась в дальнейшем, когда был пройден самый трудный этап возникновения жизни на нашей планете — когда молекулы стали «репликаторами», т. е. научились воспроизводить сами себя.
Вероятность такого события очень мала, поэтому неудивительно, что если даже некогда и существовали комплементарные пары с другими основаниями, они просто не прошли через это «бутылочное горлышко» эволюции. Впрочем, первой молекулой-репликатором, скорее всего, была не ДНК, а РНК, которая, кстати сказать, вместо тимина использует еще одно азотистое основание — урацил, также образующий пары с аденином. При переходе к «миру ДНК» урацил заменился на тимин по причинам, связанным с надежностью хранения информации.
Самое интересное, что и сейчас известны организмы, у которых «великолепная четверка» отличается от описанной в школьном учебнике. Например, у многих бактериофагов — вирусов, поражающих бактерии, место тимина в ДНК занимает урацил либо гидроксиметилурацил или другие производные урацила с дополнительно присоединенным углеводным остатком. Такая «подмена» помогает вирусу защищаться от охранных систем бактерий, расщепляющих проникшую внутрь чужеродную ДНК. А в 1970-х гг. в обыкновенной луже ленинградские микробиологи обнаружили бактериофаг, у которого аденин полностью заменен другим основанием, 2,6-диаминопурином.
Такая «подмена» помогает вирусу защищаться от охранных систем бактерий, расщепляющих проникшую внутрь чужеродную ДНК. А в 1970-х гг. в обыкновенной луже ленинградские микробиологи обнаружили бактериофаг, у которого аденин полностью заменен другим основанием, 2,6-диаминопурином.
Что касается второго вопроса, то он лег в основу нового направления молекулярной биологии — создания искусственного генетического кода. Специалисты в этой области занимаются не только поиском возможностей создания альтернативных пар оснований, но и способов введения в структуру белков неканонических аминокислот (как известно, генетические системы всех известных на сегодня живых организмов кодируют ровно 20 «стандартных» аминокислот). Понятно, что если научиться собирать ДНК из расширенного репертуара пар оснований и заложить в код возможность включения в белки нестандартных аминокислот, то это откроет невиданные перспективы перед синтетической биологией — областью науки, занимающейся созданием несуществующих в природе живых систем и процессов.
На фоне такой глобальной проблемы достижение, отмеченное журналом Science, не выглядит чем-то сногсшибательным. Скорее это — очередная ступенька лестницы, строительство которой началось два десятилетия назад, причем ступенька не очень высокая. Главный концептуальный прорыв на этом пути был совершен еще в конце 1990-х гг. группой под руководством Э. Кула (Рочестерский университет, США), которая показала, что для создания стабильной пары оснований, хорошо укладывающейся в двойную спираль ДНК, вовсе не нужны водородные связи. Можно сделать искусственные основания, вообще не содержащие ни одного атома, способного образовывать такие связи, и они смогут не только стабильно существовать в ДНК, но и без проблем включаться в нее обычными ферментами ДНК-полимеразами, по крайней мере, некоторыми из них.
В лаборатории Ф. Роумсберга, удостоенной внимания редакторов Science, неклассическими основаниями занимаются уже не первый год. Но до недавних пор все исследования в этом направлении выполнялись in vitro, т. е. «в пробирке», а не в живой клетке. В этот раз исследователи взяли одну из таких «безводородных» пар оснований и попытались заставить ее воспроизводиться внутри бактерии кишечной палочки, традиционно используемой для экспериментов молекулярными биологами.
е. «в пробирке», а не в живой клетке. В этот раз исследователи взяли одну из таких «безводородных» пар оснований и попытались заставить ее воспроизводиться внутри бактерии кишечной палочки, традиционно используемой для экспериментов молекулярными биологами.
Однако в живом организме основания не возникают по желанию экспериментатора. За каждой из четырех букв в ДНК стоит многоходовая схема их синтеза в клетке и, разумеется, основания, придуманные химиками, клетка сама делать не может. Поэтому ученые схитрили: они ввели в бактерии белок из клеточной стенки диатомовой водоросли Phaeodactylum tricornutum, который способен захватывать те самые отдельные «буквы» ДНК непосредственно из внешней среды. Соответственно, ненатуральные основания (точнее, не сами основания, а дезоксинуклеозидтрифосфаты — «кирпичики» с частью сахарофосфатного остова, из которых и строится ДНК) просто добавляли в культуральную среду, в которой росли такие бактерии.
Но существовала еще одна проблема, которую требовалось разрешить. Дело в том, что если заставить бактериальные клетки использовать ненатуральные основания в большом количестве, то бактерии просто не выживут, потому что существующий генетический аппарат не будет их «узнавать». Поэтому ученые ввели лишь одну-единственную ненатуральную пару, и то не напрямую в саму бактериальную ДНК, а в плазмиду — отдельную маленькую кольцевую молекулу ДНК, способную существовать и самовоспроизводиться внутри бактериальной клетки. А поскольку фермент ДНК-полимераза III, которая отвечает у бактерий за репликацию основной части генома, нестандартные основания вообще не узнает, ненатуральную пару оснований пришлось ставить даже не просто в плазмиду, а в очень маленький участок плазмиды, который синтезируется другим ферментом — ДНК-полимеразой I.
Дело в том, что если заставить бактериальные клетки использовать ненатуральные основания в большом количестве, то бактерии просто не выживут, потому что существующий генетический аппарат не будет их «узнавать». Поэтому ученые ввели лишь одну-единственную ненатуральную пару, и то не напрямую в саму бактериальную ДНК, а в плазмиду — отдельную маленькую кольцевую молекулу ДНК, способную существовать и самовоспроизводиться внутри бактериальной клетки. А поскольку фермент ДНК-полимераза III, которая отвечает у бактерий за репликацию основной части генома, нестандартные основания вообще не узнает, ненатуральную пару оснований пришлось ставить даже не просто в плазмиду, а в очень маленький участок плазмиды, который синтезируется другим ферментом — ДНК-полимеразой I.
После всех этих манипуляций бактерии растили на обогащенной нестандартными основаниями среде в течение 15 часов — за это время клетки успевали поделиться 24 раза. Затем определяли, что находится в том месте плазмиды, где стояла ненатуральная пара. Если бы клетка не была способна при репликации использовать соответствующие друг другу неканонические нуклеотиды, а встраивала напротив них нормальные, то ненатуральная пара после 24 делений сохранилась бы только в 1 случае из 17 млн! Однако на самом деле ничего подобного не произошло: ненатуральная пара сохранялась в 86% случаев, замена произошла только спустя нескольких суток дальнейшего роста.
Если бы клетка не была способна при репликации использовать соответствующие друг другу неканонические нуклеотиды, а встраивала напротив них нормальные, то ненатуральная пара после 24 делений сохранилась бы только в 1 случае из 17 млн! Однако на самом деле ничего подобного не произошло: ненатуральная пара сохранялась в 86% случаев, замена произошла только спустя нескольких суток дальнейшего роста.
Важность работы Роумсберга и его коллег неоспорима, ведь им действительно впервые удалось показать работоспособность ненатуральной пары оснований в живой клетке. Но говорить о том, что в ней удалось «расширить генетический алфавит», очень и очень преждевременно — эта фраза вынесена в заголовок статьи в Nature явно с рекламными целями. В конце концов, авторы статьи обошли самые главные неразрешенные проблемы искусственного генетического кода. Ведь для того, чтобы на деле расширить алфавит ДНК, нужно как минимум встроить в клетку пути синтеза неканонических нуклеотидов, сделать их совместимыми с основной системой репликации и, главное, придумать, как при помощи новых букв заставить клетку производить и новые белки.
Задача по-прежнему выглядит чрезвычайно сложной — примерно, как полет в космос в эпоху начала аэронавтики. В этом смысле работу Роумсберга и его коллег можно сравнить с запуском шара братьев Монгольфье. Но в космос в конце концов полетели не воздушные шары, так что хотя отмеченное Science достижение — безусловно, шаг в нужном направлении, пока непонятно, приведет ли к цели именно эта дорога.
Литература:
1. Власов В. В., Воробьев П. Е. Мир РНК: вчера и сегодня // Наука из первых рук. 2012. № 3(45). С. 40–49.
2. Malyshev D. A., Dhami K., Lavergne T. et al. A semi-synthetic organism with an expanded genetic alphabet // Nature. 2014. V. 509. № 7500. P. 385–388.
Расширение генетического алфавита | Национальные институты здравоохранения (NIH)
Вы находитесь здесь
Главная » Новости и события » NIH Research Matters
19 мая 2014 г.
Ученые вставили неестественную пару оснований, отмеченную X-Y, в последовательность плазмиды. исследователи, любезно предоставлено Nature
исследователи, любезно предоставлено NatureУченые создали первый живой организм, который может расти и размножаться, используя пары оснований ДНК, не встречающиеся в природе. Это достижение является важным шагом на пути к созданию новых терапевтических средств и наноматериалов.
Синтетическая биология стремится перепроектировать естественные биологические системы для новых целей. Повторно присваивая встречающиеся в природе стоп-кодоны, исследователи ранее могли включать синтетические нестандартные аминокислоты, строительные блоки белков, в белковые структуры живых клеток. Это достижение повышает вероятность того, что ученые смогут переоборудовать природу для создания новых форм белков для терапевтических и других целей.
Группа под руководством доктора Флойда Э. Ромесберга из Исследовательского института Скриппса стремилась расширить сам генетический код, тем самым обеспечив большую гибкость при создании новых биоматериалов. ДНК представляет собой длинную молекулу, состоящую из четырех химических оснований: аденина (А), гуанина (Г), цитозина (Ц) и тимина (Т). Определенные основания соединяются друг с другом — А с Т и С с G — для формирования единиц, называемых парами оснований. Порядок этих оснований определяет гены и другую информацию, необходимую клетке для функционирования.
Определенные основания соединяются друг с другом — А с Т и С с G — для формирования единиц, называемых парами оснований. Порядок этих оснований определяет гены и другую информацию, необходимую клетке для функционирования.
В лаборатории исследователи смогли расширить генетический алфавит, включив в ДНК несколько неестественных пар оснований. Но включение неестественных оснований в ДНК внутри организма создает более серьезные проблемы. Во-первых, основания доставляются в клетку белками, называемыми переносчиками нуклеотидтрифосфатов, и эти белки должны быть способны переносить неестественные основания. Во-вторых, неестественная пара оснований должна образовываться во время репликации ДНК, как естественная, и стабильно выстраиваться рядом с естественными парами в ДНК. Неестественная пара должна была бы связываться с родством, подобным родству естественных пар, чтобы разделиться и воссоединиться во время операций с ДНК. Основания также должны избегать распознавания и удаления естественными механизмами репарации ДНК.
Команда Ромесберга выбрала для этого исследования неестественную пару оснований, образованную между молекулами d5SICS и dNaM. Сначала они протестировали несколько различных переносчиков нуклеотидтрифосфатов, чтобы найти тот, который мог бы эффективно импортировать неестественные основания в клетки Escherichia coli . Их работа финансировалась Национальным институтом общих медицинских наук NIH (NIGMS). Исследование было опубликовано 15 мая 2014 года в Nature .
Команда идентифицировала переносчика из вида микроводорослей, который может импортировать неестественные основания из окружающей среды в клетку. Затем они создали штамм E. coli , из которого сделан переносчик. Наконец, они вставили в клетки плазмиду — кольцевой фрагмент ДНК, содержащий одну пару d5SICS-dNaM.
Полученная бактерия является первым организмом, способным стабильно поддерживать ДНК, состоящую из 3 типов пар оснований. Плазмида, содержащая пару d5SICS–dNaM, реплицировалась с точностью (удержание на удвоение) более 99%, что сравнимо с ошибкой репликации ДНК некоторыми природными полимеразами. Неестественная пара оснований не была удалена путями репарации ДНК, и бактерии, по-видимому, росли со скоростью, сравнимой со скоростью без синтетической системы.
Неестественная пара оснований не была удалена путями репарации ДНК, и бактерии, по-видимому, росли со скоростью, сравнимой со скоростью без синтетической системы.
«Жизнь на Земле во всем ее многообразии кодируется всего двумя парами оснований ДНК, А-Т и Ц-Г, и мы создали организм, который стабильно содержит эти две плюс третью, неестественную пару оснований», — говорит Ромесберг. . «В принципе, мы могли бы кодировать новые белки, сделанные из новых, неприродных аминокислот, что дало бы нам больше возможностей, чем когда-либо, для адаптации белковых терапевтических и диагностических и лабораторных реагентов для выполнения желаемых функций».
— Харрисон Вейн, доктор философии.
Ссылки по теме
- Ученые перекодируют геном организма
- Генетический код
- Что такое ДНК?
Каталожные номера: Полусинтетический организм с расширенным генетическим алфавитом. Малышев Д.А., Дхами К., Лавернь Т., Чен Т., Дай Н. , Фостер Дж.М., Корреа И.Р., Ромесберг Ф.Е. Природа . 15 мая 2014 г. PMID: 24805238.
, Фостер Дж.М., Корреа И.Р., Ромесберг Ф.Е. Природа . 15 мая 2014 г. PMID: 24805238.
Финансирование: Национальный институт общих медицинских наук NIH (NIGMS).
Свяжитесь с нами
- Свяжитесь с нами
- Твиттер
- Фейсбук
- Инстаграм
- Ютуб
- Flickr
ДНК получает новый — и более крупный — генетический алфавит
Наука|ДНК получает новый — и более крупный — генетический алфавит
https://www.nytimes.com/2019/02/21/science/dna-hachimoji- Genetic-alphabet.htmlРеклама
Продолжить чтение основной историиматерия
ДНК записывается четырьмя буквами или основаниями. Исследователи теперь построили систему с восемью. Это может дать ключ к пониманию потенциала жизни в других местах во Вселенной, а также расширить наши возможности по хранению цифровых данных на Земле 9.0005
Видео Анимация Милли Георгиадис/Медицинская школа Университета ИндианыКредитКредит… Милли Джорджиадис/Медицинская школа Университета Индианы
В 1985 году химик Стивен А. Беннер сел с несколькими коллегами и блокнот и набросал способ расширить алфавит ДНК. С тех пор он пытается воплотить эти наброски в реальность.
В четверг доктор Беннер и группа ученых сообщили об успехе: в статье, опубликованной в журнале Science, они заявили, что фактически удвоили генетический алфавит.
Естественная ДНК записывается четырьмя разными буквами, известными как основания — A, C, G и T. Доктор Беннер и его коллеги построили ДНК с восемью основаниями — четырьмя естественными и четырьмя неестественными. Свою новую систему они назвали Hachimoji DNA (hachi по-японски означает восемь, а moji — буква).
Создание четырех новых баз, не существующих в природе, было настоящим испытанием химической силы. Они аккуратно вписываются в двойную спираль ДНК, и ферменты могут читать их так же легко, как и природные основания, чтобы создавать молекулы.
«Здесь мы можем делать все, что необходимо для жизни», — сказал доктор Беннер, ныне заслуженный сотрудник Фонда прикладной молекулярной эволюции во Флориде.
ДНК Hachimoji может найти множество применений, в том числе гораздо более надежный способ хранения цифровых данных, который может храниться веками. «Таким образом, это может быть огромным», — сказал доктор Николас В. Хад, биохимик из Технологического института Джорджии, который не участвовал в исследованиях.
Это также поднимает глубокий вопрос о природе жизни в других частях Вселенной, предлагая возможность того, что четырехосновная ДНК, с которой мы знакомы, возможно, не единственная химия, которая может поддерживать жизнь.
[ Нравится страница Science Times на Facebook. | Подпишитесь на информационный бюллетень Science Times по номеру . ]
Все четыре естественных основания ДНК закреплены на молекулярном остове. Пара остовов может соединиться в двойную спираль, потому что их основания притягиваются друг к другу. Основания образуют связь со своими атомами водорода.
Пара остовов может соединиться в двойную спираль, потому что их основания притягиваются друг к другу. Основания образуют связь со своими атомами водорода.
Но основания не склеиваются случайным образом. C может связываться только с G, а A может связываться только с T. Эти строгие правила помогают гарантировать, что нити ДНК не слипнутся в беспорядок. Какая бы последовательность оснований не содержалась в природной ДНК, она все равно сохраняет свою форму.
Но эти четыре основания — не единственные соединения, которые могут присоединяться к остову ДНК и связываться с другим основанием — по крайней мере, на бумаге. Доктор Беннер и его коллеги придумали дюжину альтернатив.
Работая в то время в швейцарском университете ETH Zurich, доктор Беннер пытался воплотить некоторые из этих воображаемых оснований в реальность.
«Конечно, первое, что вы обнаружите, это то, что ваша теория дизайна не очень хороша», — сказал доктор Беннер.
Когда доктор Беннер и его коллеги объединили настоящие атомы, по его замыслам искусственные основания не сработали так, как он надеялся.
Тем не менее первые набеги доктора Беннера произвели впечатление на других химиков. «Его работа была для меня настоящим источником вдохновения», — сказал Флойд Э. Ромесберг, ныне работающий в Исследовательском институте Скриппса в Сан-Диего. Прочитав о ранних экспериментах доктора Беннера, доктор Ромесберг решил попробовать создать свои собственные базы.
Доктор Ромесберг решил не создавать основания, которые соединялись бы водородными связями; вместо этого он создал пару маслянистых соединений, отталкивающих воду. Эта химия объединила его неестественную пару оснований. «Нефть не любит смешиваться с водой, но ей нравится смешиваться с нефтью», — сказал доктор Ромесберг.
В последующие годы доктор Ромесберг и его коллеги создали ферменты, которые могли копировать ДНК, состоящую как из природных оснований, так и из неестественных, маслянистых. В 2014 году ученые создали бактерии, способные создавать новые копии этих гибридных генов.
В последние годы команда доктора Ромесберга начала создавать неестественные белки из этих неестественных генов.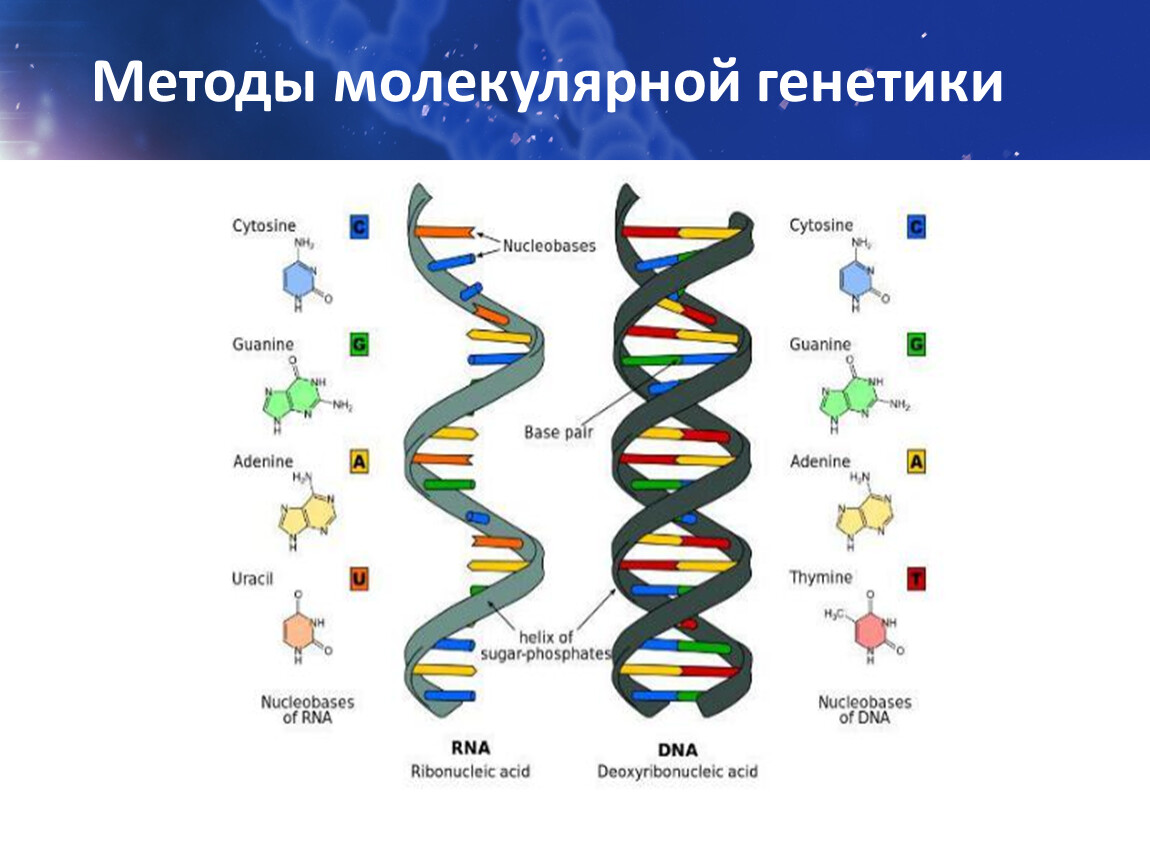 Он основал компанию Synthorx для разработки некоторых из этих белков в качестве лекарств от рака.
Он основал компанию Synthorx для разработки некоторых из этих белков в качестве лекарств от рака.
В то же время доктор Беннер продолжал свои собственные эксперименты. Ему и его коллегам удалось создать одну пару новых баз.
Как и доктор Ромесберг, они нашли применение своей неестественной ДНК. Их шестиосновная ДНК стала основой нового чувствительного теста на вирусы в образцах крови.
Затем они приступили к созданию второй пары новых баз. Теперь, имея восемь оснований для экспериментов, исследователи начали создавать молекулы ДНК с различными последовательностями. Исследователи обнаружили, что независимо от того, какую последовательность они создали, молекулы по-прежнему образуют стандартную двойную спираль.
Поскольку ДНК Хачимодзи сохраняла эту форму, она могла вести себя как обычная ДНК: она могла хранить информацию, и эта информация могла быть прочитана для создания молекулы.
Для клетки первым шагом в создании молекулы является чтение гена с помощью специальных ферментов.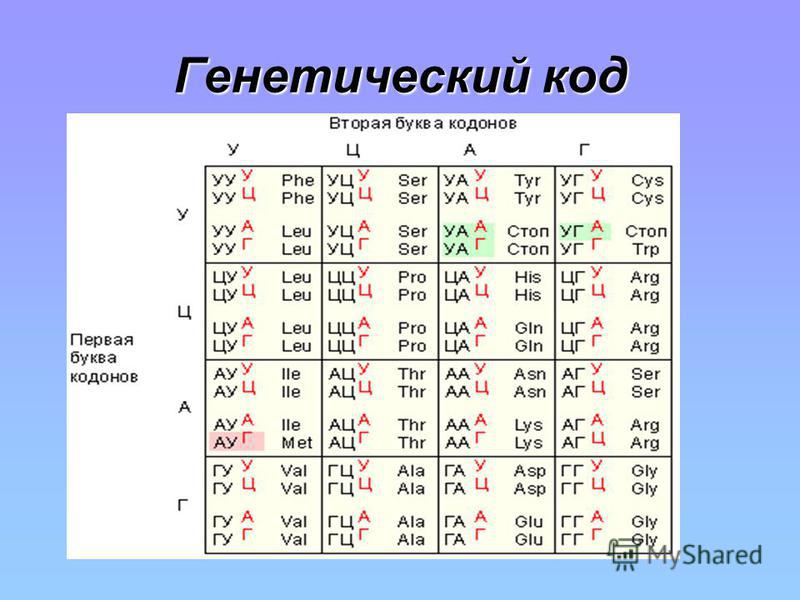 Они делают копию гена в одноцепочечной версии ДНК, называемой РНК.
Они делают копию гена в одноцепочечной версии ДНК, называемой РНК.
В зависимости от гена клетка будет делать с этой РНК одно из двух действий. В некоторых случаях он будет использовать РНК в качестве ориентира для построения белка. Но в других случаях молекула РНК уплывает, чтобы выполнить свою собственную работу.
Доктор Беннер и его коллеги создали ген Hachimoji для молекулы РНК. Они предсказали, что молекула РНК сможет захватить молекулу, называемую флуорофором. Удерживаемый молекулой РНК флуорофор поглощал свет и испускал его в виде зеленой вспышки.
Эндрю Эллингтон, инженер-эволюционист Техасского университета, руководил поиском фермента, способного читать ДНК хачимодзи. Он и его коллеги нашли многообещающий вариант, созданный вирусом, и возились с ним до тех пор, пока фермент не смог легко прочитать все восемь оснований.
Они смешали фермент в пробирках с геном Hachimoji. Как они и надеялись, их пробирки засветились зеленым.
«Вот вам все от начала до конца», — сказал доктор Беннер. «Мы можем хранить информацию, мы можем передавать ее другой молекуле, и у этой другой молекулы есть функция — и вот она, светится».
«Мы можем хранить информацию, мы можем передавать ее другой молекуле, и у этой другой молекулы есть функция — и вот она, светится».
В будущем ДНК Хатимодзи может хранить информацию совершенно другого типа. Возможно, когда-нибудь он закодирует фильм или электронную таблицу.
Сегодня фильмы, электронные таблицы и другие цифровые файлы обычно хранятся на кремниевых чипах или магнитных лентах. Но такие хранилища имеют серьезные недостатки. Во-первых, они могут испортиться всего за несколько лет.
ДНК, напротив, может оставаться неповрежденной веками. В прошлом году исследователям из Microsoft и Вашингтонского университета удалось закодировать 35 песен, видео, документов и других файлов общим объемом 200 мегабайт в пакете молекул ДНК.
Имея восемь оснований вместо четырех, ДНК Хатимодзи потенциально может кодировать гораздо больше информации. «ДНК способна хранить в два раза больше? На мой взгляд, это довольно удивительно», — сказал доктор Эллингтон.
Помимо нашей текущей потребности в хранении, ДНК Хачимодзи также предлагает некоторые подсказки о самой жизни.