Свойства и функции мотива А-минор, наиболее распространенного мотива структуры РНК – выпуск 8, том 86, 2021 – Биохимия
Финансирование
Работа не была финансирована какими-либо фондами или проектами.
Конфликт интересов
Авторы заявляют об отсутствии конфликта интересов.
Соблюдение этических норм
Настоящий обзор не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Список литературы
1. Lekka, E., and Hall, J. (2018) Noncoding RNAs in disease, FEBS Lett., 592, 2884-2900, doi: 10.1002/1873-3468.13182.
2. Novikova, I. V., Hennelly, S. P., Tung, C. S., and Sanbonmatsu, K. Y. (2013) Rise of the RNA machines: exploring the structure of long non-coding RNAs, J. Mol. Biol., 425, 3731-3746, doi: 10.1016/j.jmb.2013.02.030.
3. Leontis, N. B., Lescoute, A., and Westhof, E. (2006) The building blocks and motifs of RNA architecture, Curr. Opin. Struct. Biol., 16, 279-287, doi: 10.1016/j.sbi.2006.05.009.
Opin. Struct. Biol., 16, 279-287, doi: 10.1016/j.sbi.2006.05.009.
4. Nissen, P., Ippolito, J. A., Ban, N., Moore, P. B., and Steitz, T. A. (2001) RNA tertiary interactions in the large ribosomal subunit: the A-minor motif, Proc. Natl. Acad. Sci. USA, 98, 4899-4903, doi: 10.1073/pnas.081082398.
5. Murphy, F. L., and Cech, T. R. (1994) GAAA tetraloop and conserved bulge stabilize tertiary structure of a group I intron domain, J. Mol. Biol., 236, 49-63, doi: 10.1006/jmbi.1994.1117.
6. Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., et al. (1996) Crystal structure of a group I ribozyme domain: principles of RNA packing, Science, 273, 1678-1685, doi: 10.1126/science.273.5282.1678.
7. Scott, W. G., Finch, J. T., and Klug, A. (1995) The crystal structure of an all-RNA hammerhead ribozyme: a proposed mechanism for RNA catalytic cleavage, Cell, 81, 991-1002, doi: 10. 1016/S0092-8674(05)80004-2.
1016/S0092-8674(05)80004-2.
8. Nissen, P. (2020) The a-minor motif, in Structural Insights into Gene Expression and Protein Synthesis, pp. 461-463, doi: 10.1142/9789811215865_0055.
9. Doherty, E. A., Batey, R. T., Masquida, B., and Doudna, J. A. (2001) A universal mode of helix packing in RNA, Nat. Struct. Biol., 8, 339-343, doi: 10.1038/86221.
10. Strobel, S. A. (2002) Biochemical identification of A-minor motifs within RNA tertiary structure by interference analysis, Biochem. Soc. Transact., 30, 1126-1131, doi: 10.1042/bst0301126.
11. Krasilnikov, A. S., Yang, X., Pan, T., and Mondragón, A. (2003) Crystal structure of the specificity domain of ribonuclease P, Nature, 421, 760-764, doi: 10.1038/nature01386.
12. Krasilnikov, A. S., Xiao, Y., Pan, T., and Mondragón, A. (2004) Basis for structural diversity in homologous RNAs, Science, 306, 104-107, doi: 10. 1126/science.1101489.
1126/science.1101489.
13. Nagai, K., Oubridge, C., Kuglstatter, A., Menichelli, E., Isel, C., and Jovine, L. (2003) Structure, function and evolution of the signal recognition particle, EMBO J., 22, 3479-3485, doi: 10.1093/emboj/cdg337.
14. Schüler, M., Connell, S. R., Lescoute, A., Giesebrecht, J., Dabrowski, M., et al. (2006) Structure of the ribosome-bound cricket paralysis virus IRES RNA, Nat. Struct. Mol. Biol., 13, 1092-1096, doi: 10.1038/nsmb1177.
15. Mitton-Fry, R. M., DeGregorio, S. J., Wang, J., Steitz, T. A., and Steitz, J. A. (2010) Poly (A) tail recognition by a viral RNA element through assembly of a triple helix, Science, 330, 1244-1247, doi: 10.1126/science.1195858.
16. Nguyen, L. A., Wang, J., and Steitz, T. A. (2017) Crystal structure of Pistol, a class of self-cleaving ribozyme, Proc. Natl. Acad. Sci. USA, 114, 1021-1026, doi: 10.1073/pnas.1611191114.
17. Xue, S., Calvin, K., and Li, H. (2006) RNA recognition and cleavage by a splicing endonuclease, Science, 312, 906-910, doi: 10.1126/science.1126629.
18. Serganov, A., Yuan, Y. R., Pikovskaya, O., Polonskaia, A., Malinina, L., et al. (2004) Structural basis for discriminative regulation of gene expression by adenine-and guanine-sensing mRNAs, Chem. Biol., 11, 1729-1741, doi: 10.1016/j.chembiol.2004.11.018.
19. Dann, C. E. 3rd, Wakeman, C. A., Sieling, C. L., Baker, S. C., Irnov, I., and Winkler, W. C. (2007) Structure and mechanism of a metal-sensing regulatory RNA, Cell, 130, 878-892, doi: 10.1016/j.cell.2007.06.051.
20. Jones, C. P., and Ferré-D’Amaré, A. R. (2015) RNA quaternary structure and global symmetry, Trends Biochem. Sci., 40, 211-220, doi: 10.1016/j.tibs.2015.02.004.
21. Brown, J. A., Bulkley, D., Wang, J., Valenstein, M. L., Yario, T. A., et al. (2014) Structural insights into the stabilization of MALAT1 noncoding RNA by a bipartite triple helix, Nat. Struct. Mol. Biol., 21, 633, doi: 10.1038/nsmb.2844.
(2014) Structural insights into the stabilization of MALAT1 noncoding RNA by a bipartite triple helix, Nat. Struct. Mol. Biol., 21, 633, doi: 10.1038/nsmb.2844.
22. Klein, D. J., Schmeing, T. M., Moore, P. B., and Steitz, T. A. (2001) The kink-turn: a new RNA secondary structure motif, EMBO J., 20, 4214-4221, doi: 10.1093/emboj/20.15.4214.
23. Réblová, K., Šponer, J. E., Špačková, N., Beššeová, I., and Šponer, J. (2011) A-minor tertiary interactions in RNA kink-turns. Molecular dynamics and quantum chemical analysis, J. Phys. Chem. B, 115, 13897-13910, doi: 10.1021/jp2065584.
24. Geary, C., Baudrey, S., and Jaeger, L. (2008) Comprehensive features of natural and in vitro selected GNRA tetraloop-binding receptors, Nucleic Acids Res., 36, 1138-1152 10.1093/nar/gkm1048.
25. Wu, L., Chai, D., Fraser, M. E., and Zimmerly, S. (2012) Structural variation and uniformity among tetraloop-receptor interactions and other loop-helix interactions in RNA crystal structures, PLoS One, 7, e49225, doi: 10. 1371/journal.pone.0049225.
1371/journal.pone.0049225.
26. Fiore, J. L., and Nesbitt, D. J. (2013) An RNA folding motif: GNRA tetraloop–receptor interactions, Quart. Rev. Biophys., 46, 223-264, doi: 10.1017/S0033583513000048.
27. Aalberts, D. P., and Hodas, N. O. (2005) Asymmetry in RNA pseudoknots: observation and theory, Nucleic Acids Res., 33, 2210-2214, doi: 10.1093/nar/gki508.
28. Giedroc, D. P., and Cornish, P. V. (2009) Frameshifting RNA pseudoknots: structure and mechanism, Virus Res., 139, 193-208, doi: 10.1016/j.virusres.2008.06.008.
29. Lescoute, A., and Westhof, E. (2006) Topology of three-way junctions in folded RNAs, RNA, 12, 83-93, doi: 10.1261/rna.2208106.
30. Xin, Y., Laing, C., Leontis, N. B., and Schlick, T. (2008) Annotation of tertiary interactions in RNA structures reveals variations and correlations, RNA, 14, 2465-2477, doi: 10.1261/rna. 1249208.
1249208.
31. Laing, C., and Schlick, T. (2009) Analysis of four-way junctions in RNA structures, J. Mol. Biol., 390, 547-559, doi: 10.1016/j.jmb.2009.04.084.
32. Cruz, J. A., and Westhof, E. (2009) The dynamic landscapes of RNA architecture, Cell, 136, 604-609, doi: 10.1016/j.cell.2009.02.003.
33. Geary, C., Chworos, A., and Jaeger, L. (2011) Promoting RNA helical stacking via A-minor junctions, Nucleic Acids Res., 39, 1066-1080, doi: 10.1093/nar/gkq748.
34. Tamura, M., and Holbrook, S. R. (2002) Sequence and structural conservation in RNA ribose zippers, J. Mol. Biol., 320, 455-474, doi: 10.1016/S0022-2836(02)00515-6.
35. Lee, J. C., Cannone, J. J., and Gutell, R. R. (2003) The lonepair triloop: a new motif in RNA structure, J. Mol. Biol., 325, 65-83, doi: 10.1016/S0022-2836(02)01106-3.
36. Gagnon, M. G., and Steinberg, S. V. (2010) The adenosine wedge: A new structural motif in ribosomal RNA, RNA, 16, 375-381, doi: 10.1261/rna.1550310.
V. (2010) The adenosine wedge: A new structural motif in ribosomal RNA, RNA, 16, 375-381, doi: 10.1261/rna.1550310.
37. Leontis, N. B., and Westhof, E. (2001) Geometric nomenclature and classification of RNA base pairs, RNA, 7, 499-512, doi: 10.1017/s1355838201002515.
38. Torabi, S. F., Vaidya, A. T., Tycowski, K. T., DeGregorio, S. J., Wang, J., et al. (2021) RNA stabilization by a poly (A) tail 3′-end binding pocket and other modes of poly (A)-RNA interaction, Science, 371, eabe6523, doi: 10.1126/science.abe6523.
39. Newby, M. I., and Greenbaum, N. L. (2002) Sculpting of the spliceosomal branch site recognition motif by a conserved pseudouridine, Nat. Struct. Biol., 9, 958-965, doi: 10.1038/nsb873.
40. Hamdani, H. Y., and Firdaus-Raih, M. (2019) Identification of structural motifs using networks of hydrogen-bonded base interactions in RNA crystallographic structures, Crystals, 9, 550, doi: 10. 3390/cryst9110550.
3390/cryst9110550.
41. Lescoute, A., and Westhof, E. (2006) The A-minor motifs in the decoding recognition process, Biochimie, 88, 993-999, doi: 10.1016/j.biochi.2006.05.018.
42. Lescoute, A., and Westhof, E. (2006) The interaction networks of structured RNAs, Nucleic Acids Res., 34, 6587-6604, doi: 10.1093/nar/gkl963.
43. Petrov, A. I., Zirbel, C. L., and Leontis, N. B. (2011) WebFR3D – a server for finding, aligning and analyzing recurrent RNA 3D motifs, Nucleic Acids Res., 39, W50-W55, doi: 10.1093/nar/gkr249.
44. Sheth, P., Cervantes-Cervantes, M., Nagula, A., Laing, C., and Wang, J. T. (2013) Novel features for identifying A-minors in three-dimensional RNA molecules, Computat. Biol. Chem., 47, 240-245, doi: 10.1016/j.compbiolchem.2013.10.004.
45. Laing, C., Jung, S., Iqbal, A., and Schlick, T. (2009) Tertiary motifs revealed in analyses of higher-order RNA junctions, J. Mol. Biol., 393, 67-82, doi: 10.1016/j.jmb.2009.07.089.
Mol. Biol., 393, 67-82, doi: 10.1016/j.jmb.2009.07.089.
46. Burley, S. K., Berman, H. M., Kleywegt, G. J., Markley, J. L., Nakamura, H., and Velankar, S. (2017) Protein Data Bank (PDB): the single global macromolecular structure archive, Protein Crystallogr., 1607, 627-641, doi: 10.1007/978-1-4939-7000-1_26.
47. Reinharz, V., Soulé, A., Westhof, E., Waldispühl, J., and Denise, A. (2018) Mining for recurrent long-range interactions in RNA structures reveals embedded hierarchies in network families, Nucleic Acids Res., 46, 3841-3851, doi: 10.1093/nar/gky197.
48. Appasamy, S. D., Hamdani, H. Y., Ramlan, E. I., and Firdaus-Raih, M. (2016) InterRNA: a database of base interactions in RNA structures, Nucleic Acids Res., 44, D266-D271, doi: 10.1093/nar/gkv1186.
49. Hamdani, H. Y., Appasamy, S. D., Willett, P., Artymiuk, P. J., and Firdaus-Raih, M. (2012) NASSAM: a server to search for and annotate tertiary interactions and motifs in three-dimensional structures of complex RNA molecules, Nucleic Acids Res. , 40, W35-W41, doi: 10.1093/nar/gks513.
, 40, W35-W41, doi: 10.1093/nar/gks513.
50. Firdaus-Raih, M., Hamdani, H. Y., Nadzirin, N., Ramlan, E. I., Willett, P., and Artymiuk, P. J. (2014) COGNAC: a web server for searching and annotating hydrogen-bonded base interactions in RNA three-dimensional structures, Nucleic Acids Res., 42, W382-W388, doi: 10.1093/nar/gku438.
51. Lu, X. J., Bussemaker, H. J., and Olson, W. K. (2015) DSSR: an integrated software tool for dissecting the spatial structure of RNA, Nucleic Acids Res., 43, e142-e142, doi: 10.1093/nar/gkv716.
52. Yang, H., Jossinet, F., Leontis, N., Chen, L., Westbrook, J., et al. (2003) Tools for the automatic identification and classification of RNA base pairs, Nucleic Acids Res., 31, 3450-3460, doi: 10.1093/nar/gkg529.
53. Gendron, P., Lemieux, S., and Major, F. (2001) Quantitative analysis of nucleic acid three-dimensional structures, J. Mol. Biol.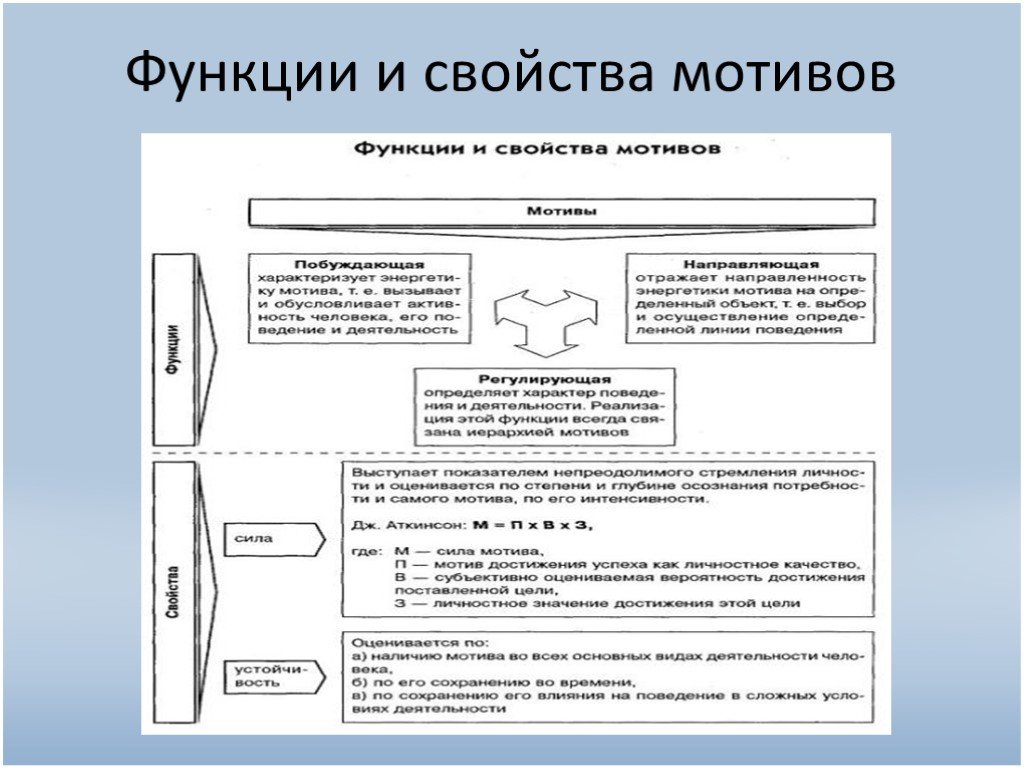 , 308, 919-936, doi: 10.1006/jmbi.2001.4626.
, 308, 919-936, doi: 10.1006/jmbi.2001.4626.
54. Lu, X. J., and Olson, W. K. (2008) 3DNA: a versatile, integrated software system for the analysis, rebuilding and visualization of three-dimensional nucleic-acid structures, Nat. Protoc., 3, 1213, doi: 10.1038/nprot.2008.104.
55. Shalybkova, A. A., Mikhailova, D. S., Kulakovskiy, I. V., Fakhranurova, L. I., and Baulin, E. F. (2021) Annotation of the local context of the RNA secondary structure improves the classification and prediction of A-minors, RNA, rna-078535, doi: 10.1261/rna.078535.120.
56. Rázga, F., Koča, J., Šponer, J., and Leontis, N. B. (2005) Hinge-like motions in RNA kink-turns: the role of the second A-minor motif and nominally unpaired bases, Biophys. J., 88, 3466-3485, doi: 10.1529/biophysj.104.054916.
57. Sponer, J., Bussi, G., Krepl, M., Banaš, P., Bottaro, S., et al. (2018) RNA structural dynamics as captured by molecular simulations: a comprehensive overview, Chem. Rev., 118, 4177-4338, doi: 10.1021/acs.chemrev.7b00427.
Rev., 118, 4177-4338, doi: 10.1021/acs.chemrev.7b00427.
58. Laing, C., Wen, D., Wang, J. T., and Schlick, T. (2012) Predicting coaxial helical stacking in RNA junctions, Nucleic Acids Res., 40, 487-498, doi: 10.1093/nar/gkr629.
59. Beššeová, I., Reblova, K., Leontis, N. B., and Šponer, J. (2010) Molecular dynamics simulations suggest that RNA three-way junctions can act as flexible RNA structural elements in the ribosome, Nucleic Acids Res., 38, 6247-6264, doi: 10.1093/nar/gkq414.
60. Lescoute, A., and Westhof, E. (2005) Riboswitch structures: purine ligands replace tertiary contacts, Chem. Biol., 12, 10-13, doi: 10.1016/j.chembiol.2005.01.002.
61. Baulin, E., Yacovlev, V., Khachko, D., Spirin, S., and Roytberg, M. (2016) URS DataBase: universe of RNA structures and their motifs, Database, 2016, baw085, doi: 10.1093/database/baw085.
62. Calvin, K., and Li, H. (2008) RNA-splicing endonuclease structure and function, Cell. Mol. Life Sci., 65, 1176-1185, doi: 10.1007/s00018-008-7393-y.
Calvin, K., and Li, H. (2008) RNA-splicing endonuclease structure and function, Cell. Mol. Life Sci., 65, 1176-1185, doi: 10.1007/s00018-008-7393-y.
63. Ikawa, Y., Yoshimura, T., Hara, H., Shiraishi, H., and Inoue, T. (2002) Two conserved structural components, A-rich bulge and P4 XJ6/7 base-triples, in activating the group I ribozymes, Genes Cells, 7, 1205-1215, doi: 10.1046/j.1365-2443.2002.00601.x.
64. Battle, D. J., and Doudna, J. A. (2002) Specificity of RNA–RNA helix recognition, Proc. Natl. Acad. Sci. USA, 99, 11676-11681, doi: 10.1073/pnas.182221799.
65. Schwalbe, H., Buck, J., Fürtig, B., Noeske, J., and Wöhnert, J. (2007) Structures of RNA switches: insight into molecular recognition and tertiary structure, Angewandte Chemie Int. Edn., 46, 1212-1219, doi: 10.1002/anie.200604163.
66. Šponer, J. E., Leszczynski, J., Sychrovský, V., and Šponer, J. (2005) Sugar edge/sugar edge base pairs in RNA: stabilities and structures from quantum chemical calculations, J. Phys. Chem. B, 109, 18680-18689, doi: 10.1021/jp053379q.
Phys. Chem. B, 109, 18680-18689, doi: 10.1021/jp053379q.
67. Šponer, J. E., Reblova, K., Mokdad, A., Sychrovský, V., Leszczynski, J., and Šponer, J. (2007) Leading RNA tertiary interactions: structures, energies, and water insertion of A-minor and P-interactions. A quantum chemical view, J. Phys. Chem. B, 111, 9153-9164, doi: 10.1021/jp0704261.
68. Costa, M., and Michel, F. (1995) Frequent use of the same tertiary motif by self-folding RNAs, EMBO J., 14, 1276-1285, doi: 10.1002/j.1460-2075.1995.tb07111.x.
69. Lee, J. C., Gutell, R. R., and Russell, R. (2006) The UAA/GAN internal loop motif: a new RNA structural element that forms a cross-strand AAA stack and long-range tertiary interactions, J. Mol. Biol., 360, 978-988, doi: 10.1016/j.jmb.2006.05.066.
70. Yoshizawa, S., Fourmy, D., and Puglisi, J. D. (1999) Recognition of the codon-anticodon helix by ribosomal RNA, Science, 285, 1722-1725, doi: 10. 1126/science.285.5434.1722.
1126/science.285.5434.1722.
71. Ogle, J. M., Brodersen, D. E., Clemons, W. M., Tarry, M. J., Carter, A. P., and Ramakrishnan, V. (2001) Recognition of cognate transfer RNA by the 30S ribosomal subunit, Science, 292, 897-902, doi: 10.1126/science.1060612.
72. Ogle, J. M., and Ramakrishnan, V. (2005) Structural insights into translational fidelity, Annu. Rev. Biochem., 74, 129-177, doi: 10.1146/annurev.biochem.74.061903.155440.
73. Gromadski, K. B., Daviter, T., and Rodnina, M. V. (2006) A uniform response to mismatches in codon-anticodon complexes ensures ribosomal fidelity, Mol. Cell, 21, 369-377, doi: 10.1016/j.molcel.2005.12.018.
74. Prokhorova, I., Altman, R. B., Djumagulov, M., Shrestha, J. P., Urzhumtsev, A., et al. (2017) Aminoglycoside interactions and impacts on the eukaryotic ribosome, Proc. Natl. Acad. Sci. USA, 114, E10899-E10908, doi: 10.1073/pnas.1715501114.
75. Steitz, T. A., and Moore, P. B. (2003) RNA, the first macromolecular catalyst: the ribosome is a ribozyme, Trends Biochem. Sci., 28, 411-418, doi: 10.1016/S0968-0004(03)00169-5.
76. Noller, H. F. (2012) Evolution of protein synthesis from an RNA world, Cold Spring Harb. Perspect. Biol., 4, a003681, doi: 10.1101/cshperspect.a003681.
77. Lancaster, L., and Noller, H. F. (2005) Involvement of 16S rRNA nucleotides G1338 and A1339 in discrimination of initiator tRNA, Mol. Cell, 20, 623-632, doi: 10.1016/j.molcel.2005.10.006.
78. Steitz, T. A. (2008) A structural understanding of the dynamic ribosome machine, Nat. Rev. Mol. Cell Biol., 9, 242-253, doi: 10.1038/nrm2352.
79. Hansen, J. L., Schmeing, T. M., Moore, P. B., and Steitz, T. A. (2002) Structural insights into peptide bond formation, Proc. Natl. Acad. Sci. USA, 99, 11670-11675, doi: 10. 1073/pnas.172404099.
1073/pnas.172404099.
80. Noller, H. F. (2005) RNA structure: reading the ribosome, Science, 309, 1508-1514, doi: 10.1126/science.1111771.
81. Szymański, M., Barciszewska, M. Z., Erdmann, V. A., and Barciszewski, J. (2003) 5S rRNA: structure and interactions, Biochem. J., 371, 641-651, doi: 10.1042/bj20020872.
82. Mohan, S., and Noller, H. F. (2017) Recurring RNA structural motifs underlie the mechanics of L1 stalk movement, Nat. Commun., 8, 1-11, doi: 10.1038/ncomms14285.
83. Bou-Nader, C., and Zhang, J. (2020) Structural insights into RNA dimerization: Motifs, interfaces and functions, Molecules, 25, 2881, doi: 10.3390/molecules25122881.
84. Leontis, N. B., and Westhof, E. (2003) Analysis of RNA motifs, Curr. Opin. Struct. Biol., 13, 300-308, doi: 10.1016/S0959-440X(03)00076-9.
85. Frank, J., Gao, H., Sengupta, J., Gao, N. , and Taylor, D. J. (2007) The process of mRNA–tRNA translocation, Proc.Natl. Acad. Sci. USA, 104, 19671-19678, doi: 10.1073/pnas.0708517104.
, and Taylor, D. J. (2007) The process of mRNA–tRNA translocation, Proc.Natl. Acad. Sci. USA, 104, 19671-19678, doi: 10.1073/pnas.0708517104.
86. Spirin, A. S. (1968) How does the ribosome work? A hypothesis based on the two subunit construction of the ribosome, Curr. Mod. Biol., 2, 115-127, doi: 10.1016/0303-2647(68)90017-8.
Перечень всех учебных материаловГосударство и правоДемография История Международные отношения Педагогика Политические науки Психология Религиоведение Социология |
§ 1.3.1. Структура и функции мотива Существовали попытки отождествить мотив с каким-либо отдельно взятым психологическим явлением, например, с потребностью, целью, состоянием или свойствами личности. Однако, ряд авторов (например, В. Рис. 6 Перечень компонентов, которые могут создавать структуру мотива В конкретном случае в каждом блоке может быть взят в качестве основания действия или поступка один из компонентов. Структура же каждого конкретного мотива строится из сочетания тех компонентов, которые обусловили принятое человеком решение. Набор компонентов в каждом конкретном мотиве может быть разным. Но и сходство внешней структуры мотива у разных лиц не означает их тождества по содержанию. Что касается функций мотива, то в качестве основных можно выделить побуждающую и направляющую функции. Первая отражает энергетику, вторая — направленность этой энергии на цель. По мере продвижения к цели энергетический потенциал, который призван обеспечить достижение целевого объекта, часто падает, в связи с чем в ряде случаев для окончания действия требуется дополнительная волевая стимуляция. С этим явлением связывают стимулирующую функцию мотива. Ряд авторов (М. Ш. Магомед-Эминов, П. А. Рудик) предлагают объединить первую и вторую функции и назвать результат пусковой функцией (или директивной, по П. А. Рудику). Однако при этом, по их мнению, за пределами внимания остается регулятивная функция, являющаяся центральной в процессах мотивации. Можно говорить также об управляющей, организующей, структурирующей функциях мотива. Мы уже отмечали, что одна из наиболее разработанных концепций мотивации в отечественной психологии принадлежит А. Н. Леонтьеву. Данный исследователь выделял смыслообразующую функцию мотива, предполагая, что мотив придает личностный смысл действиям и определенным обстоятельствам.  Он писал, что мотивы не «отделены» от сознания. Даже когда мотивы не сознаются субъектом, т. е. когда он не отдает себе отчета в том, что побуждает его осуществлять ту или иную деятельность, они, образно говоря, входят в его сознание, но только особым образом. Они придают сознательному отражению субъективную окрашенность, которая выражает значение отражаемого для самого субъекта, его, как мы говорим, личностный смысл. Таким образом, кроме своей основной функции-функции побуждения, мотивы имеют еще и вторую функцию — функцию смыслообразования. Следует отметить, что обоснование А. Н. Леонтьевым смыслообразующей функции мотива не безупречно, в связи с чем ее наличие рядом авторов отрицается. Например, В. И. Ковалев считает выделение данной функции нецелесообразным и малообоснованным, поскольку личностный смысл относится к самой сущности мотива. Мы предлагаем сохранить указанную функцию за мотивом. Он писал, что мотивы не «отделены» от сознания. Даже когда мотивы не сознаются субъектом, т. е. когда он не отдает себе отчета в том, что побуждает его осуществлять ту или иную деятельность, они, образно говоря, входят в его сознание, но только особым образом. Они придают сознательному отражению субъективную окрашенность, которая выражает значение отражаемого для самого субъекта, его, как мы говорим, личностный смысл. Таким образом, кроме своей основной функции-функции побуждения, мотивы имеют еще и вторую функцию — функцию смыслообразования. Следует отметить, что обоснование А. Н. Леонтьевым смыслообразующей функции мотива не безупречно, в связи с чем ее наличие рядом авторов отрицается. Например, В. И. Ковалев считает выделение данной функции нецелесообразным и малообоснованным, поскольку личностный смысл относится к самой сущности мотива. Мы предлагаем сохранить указанную функцию за мотивом. Данная функция представляет отражение в сознании человека потребностей и целей, средств их достижения и своих возможностей. Мотивация, с этой точки зрения, отражает все предшествовавшие влияния социальной среды. Данная функция представляет отражение в сознании человека потребностей и целей, средств их достижения и своих возможностей. Мотивация, с этой точки зрения, отражает все предшествовавшие влияния социальной среды.Наконец, К. Обуховский говорит о защитной функции мотива, которая предполагает подмену истинного объяснения поведения «официальной версией», необходимой для сохранения требуемого решения, для создания видимости рациональной деятельности. В связи с этим выделяют такой мотивационный феномен, как мотивировка. Мотивировка определяется как рациональное объяснение субъектом причин действия посредством указания на социально приемлемые для него и референтной группы обстоятельства, побудившие к выбору данного действия. С помощью мотивировок личность иногда оправдывает свои действия и поступки, приводя их в соответствие с нормами поведения в обществе и со своими личностными нормативами. Возможным представляется использование мотивировок для сохранения Я-концепции.
|
||||
|
© www. |
Понимание и оценка мотивации волонтеров: функциональный подход
. 1998 июнь; 74 (6): 1516-30. doi: 10.1037//0022-3514.74.6.1516.Э Г Клэри 1 , M Snyder, R D Ridge, J Copeland, A A Stukas, J Haugen, P Miene
принадлежность
- 1 Факультет психологии, Колледж Святой Екатерины, Сент-Пол, Миннесота 55105, США. [email protected]
- PMID: 9654757
- DOI: 10.1037//0022-3514.74.6.1516
EG Clary et al.
J Pers Soc Psychol. 1998 июнь
1998 июнь
Авторы
Э Г Клэри 1 , М. Снайдер, Р. Д. Ридж, Дж. Коупленд, А. А. Стукас, Дж. Хауген, П. Миене
принадлежность
- 1 Факультет психологии, Колледж Святой Екатерины, Сент-Пол, Миннесота 55105, США. [email protected]
- PMID: 9654757
- DOI: 10.1037//0022-3514.74.6.1516
Абстрактный
Авторы применили функционалистскую теорию к вопросу о мотивах, лежащих в основе волонтерства, выдвинули гипотезу о шести функциях, потенциально выполняемых волонтерством, и разработали инструмент для оценки этих функций (опросник волонтерских функций; VFI).
Похожие статьи
- Перевод и валидация перечня волонтерских функций (VFI) среди пожилого населения Нидерландов в целом.

Нибур Дж., Лифброер А.С., Стеверинк Н., Смидт Н. Нибур Дж. и др. Общественное здравоохранение Int J Environ Res. 2019 авг 26;16(17):3106. дои: 10.3390/ijerph26173106. Общественное здравоохранение Int J Environ Res. 2019. PMID: 31455027 Бесплатная статья ЧВК.
- Мотивы добровольцев: функциональная стратегия набора, размещения и удержания добровольцев.
Клэри Э.Г., Снайдер М., Ридж Р. Клэри Э.Г. и др. Некоммерческая организация Manag Leadersh. Лето 1992 г .; 2 (4): 333–50. doi: 10.1002/nml.4130020403. Некоммерческая организация Manag Leadersh. 1992. PMID: 10119110
- Практические последствия понимания влияния мотивов на приверженность добровольному управлению сохранением городов.
Asah ST, Blahna DJ.
 Асах С.Т. и др.
Консерв Биол. 2013 авг; 27 (4): 866-75. doi: 10.1111/cobi.12058. Epub 2013 8 мая.
Консерв Биол. 2013.
PMID: 23656329
Асах С.Т. и др.
Консерв Биол. 2013 авг; 27 (4): 866-75. doi: 10.1111/cobi.12058. Epub 2013 8 мая.
Консерв Биол. 2013.
PMID: 23656329 - Волонтеры по борьбе со СПИДом и их мотивация: теоретические вопросы и практические проблемы.
Омото А.М., Снайдер М. Омото А.М. и др. Некоммерческая организация Manag Leadersh. 1993 Зима; 4 (2): 157-76. doi: 10.1002/nml.4130040204. Некоммерческая организация Manag Leadersh. 1993. PMID: 10133035 Обзор.
- Возраст и мотивы волонтерства: проверка гипотез, вытекающих из теории социально-эмоциональной избирательности.
Окунь М.А., Шульц А. Окунь М.А. и соавт. Психологическое старение. 2003 июнь; 18 (2): 231-9. дои: 10.1037/0882-7974.18.2.231. Психологическое старение.
 2003.
PMID: 12825773
Обзор.
2003.
PMID: 12825773
Обзор.
Посмотреть все похожие статьи
Цитируется
- Понимание мотивов и проблем добровольцев в тренерской и судейской работе в баскетболе: последствия для спортивной политики.
Кейси М., Харви Дж., Чарити М., Талпи С., Рис Л., Эйме Р. Кейси М. и др. Общественное здравоохранение BMC. 2023 25 мая; 23(1):946. doi: 10.1186/s12889-023-15949-5. Общественное здравоохранение BMC. 2023. PMID: 37231371 Бесплатная статья ЧВК.
- Благотворительные пожертвования и теория запланированного поведения: систематический обзор и метаанализ.
Уайт К.М., Старфелт Саттон Л.С., Чжао Х. Уайт К.М. и др. ПЛОС Один. 2023 19 мая; 18 (5): e0286053. doi: 10.1371/journal.
 pone.0286053. Электронная коллекция 2023.
ПЛОС Один. 2023.
PMID: 37205662
Бесплатная статья ЧВК.
pone.0286053. Электронная коллекция 2023.
ПЛОС Один. 2023.
PMID: 37205662
Бесплатная статья ЧВК. - Изучение опыта волонтеров-помощников по выращиванию щенков собак из одной и той же программы в двух кампусах австралийских университетов.
Морвуд С., Май Д., Беннет П.С., Бентон П., Хауэлл Т.Дж. Морвуд С. и др. Животные (Базель). 2023 27 апреля; 13 (9): 1482. дои: 10.3390/ани13091482. Животные (Базель). 2023. PMID: 37174519 Бесплатная статья ЧВК.
- ПРОТОКОЛ: Добровольная работа для физического и психического здоровья пожилых добровольцев.
Филгес Т., Сирен А., Фридберг Т., Кристиан Б., Нильсен В. Филгес Т. и др. Campbell Syst Rev. 27 ноября 2018 г .; 14 (1): 1–40. дои: 10.1002/CL2.190. Электронная коллекция 2018. Кэмпбелл Сист Ред.
 2018.
PMID: 37131403
Бесплатная статья ЧВК.
Аннотация недоступна.
2018.
PMID: 37131403
Бесплатная статья ЧВК.
Аннотация недоступна. - Добровольная работа для физического и психического здоровья пожилых добровольцев: систематический обзор.
Филгес Т., Сирена А., Фридберг Т., Нильсен BCV. Филгес Т. и др. Campbell Syst Rev. 2020 Oct 23;16(4):e1124. doi: 10.1002/cl2.1124. Электронная коллекция 2020 декабрь. Кэмпбелл Сист Ред. 2020. PMID: 37016617 Бесплатная статья ЧВК. Обзор.
Просмотреть все статьи «Цитируется по»
Типы публикаций
термины MeSH
Мозговые цепи, лежащие в основе мотивации: интерактивная графика
Инфографика
Щелкните любую из стрелок или кнопок выше, чтобы просмотреть дополнительную информацию. Посмотреть увеличенную версию рисунка. (Полезно для мобильных устройств.)
Посмотреть увеличенную версию рисунка. (Полезно для мобильных устройств.)
Что такое мотивация? Системы мозга, управляющие мотивацией, формируются с течением времени, начиная с самых ранних лет развития. Эти сложные нейронные цепи и структуры формируются в результате взаимодействия между нашим опытом и генами, с которыми мы рождаемся, которые вместе влияют как на то, как развиваются наши системы мотивации, так и на то, как они функционируют в дальнейшей жизни.
На интерактивном графике выше наведите указатель мыши на метки областей мозга или щелкните их, чтобы узнать больше о том, как каждая область влияет на мотивацию, и наведите указатель мыши или щелкните выделенный текст слева от изображения мозга, чтобы увидеть, как взаимодействуют эти области.
Этот рисунок взят из рабочего документа № 14 Национального научного совета по развитию ребенка «Понимание мотивации: построение архитектуры мозга, поддерживающей обучение, здоровье и участие в жизни общества».
Иллюстрация мозга Бетси Хейс
Полный текст рисунка
Опыт создает пути между областями мозга: как развиваются системы мотивации
На графике показано изображение человеческого мозга, наложенное на профиль головы человека. Маркированы различные области мозга, в том числе:
- Миндалина
- «Триггер эмоций» — быстро оценивает поступающую информацию из окружающей среды и активирует поведение приближения или избегания. Эта структура имеет решающее значение для обнаружения угроз и выученного страха.
- Пути дофамина
- Дофамин, ключевой фактор «желания», модулирует нейронную активность, когда происходит полезное событие. Повышение уровня дофамина усиливает поведение, вызывающее вознаграждение, и побуждает людей искать и учиться на новом опыте в ожидании положительного результата.
- Гиппокамп
- «Центр памяти» — записывает подробные воспоминания о событиях и вызывает извлечение этих воспоминаний при подаче соответствующей подсказки.
 Эта структура также участвует в регуляции продолжительности стрессовых реакций на раздражители окружающей среды.
Эта структура также участвует в регуляции продолжительности стрессовых реакций на раздражители окружающей среды.
- «Центр памяти» — записывает подробные воспоминания о событиях и вызывает извлечение этих воспоминаний при подаче соответствующей подсказки.
- Передняя поясная кора (ACC)
- «Отслеживание поведения» — отслеживает окружающую среду, а также собственное поведение и поведение других (например, социальную изоляцию). Эта область подает сигнал тревоги, когда необходимо изменить поведение, мобилизуя участки префронтальной коры, участвующие в саморегуляции и принятии решений.
- Префронтальная кора
- «Управление воздушным движением» — управляет исполнительными функциями, саморегуляцией, контролем поведения, планированием и принятием сложных решений.
- Прилежащее ядро
- «Предвосхищающий вознаграждение» — оценивает стимулы, вызывающие реакцию желания или симпатии. Эта структура также играет важную роль в обучении на основе обратной связи и в принятии решений на основе вознаграждения.
- Черная субстанция/вентральная область покрышки (VTA)
- «Распределители дофамина» — вырабатывают дофамин в мозгу и доставляют его в другие области мозга, которые участвуют в двигательной функции, а также в мотивации и поощрении поведения.

- «Распределители дофамина» — вырабатывают дофамин в мозгу и доставляют его в другие области мозга, которые участвуют в двигательной функции, а также в мотивации и поощрении поведения.
- Ядра шва
- «Распределители серотонина» — производят нейротрансмиттер серотонин и доставляют его в широкую сеть контуров мозга, включая структуры, связанные с мотивацией, вознаграждением,
и обнаружением угроз.
- «Распределители серотонина» — производят нейротрансмиттер серотонин и доставляют его в широкую сеть контуров мозга, включая структуры, связанные с мотивацией, вознаграждением,
- Пути серотонина
- Ключевой фактор «приязни», серотонин в сочетании с другими нейрохимическими веществами вызывает эйфорию и имеет самое широкое распространение в мозгу. Серотонин модулирует широкий спектр поведения, в том числе в качестве основного влияния на эмоциональные состояния, циклы сна, прием пищи и другие полезные действия.
В левой части изображения мозга перечислены способы развития систем мотивации:
- Опыт запускает нейроны (клетки головного мозга) в определенных областях мозга, включая префронтальную кору, переднюю поясную кору и гиппокамп, для отправки химических веществ, таких как дофамин и серотонин, в другие нейроны в разных областях.
- Повторяющиеся переживания создают в мозгу различные пути, например, для дофамина и серотонина, которые связывают эти переживания с мыслями, воспоминаниями (в гиппокампе) и поведением.

 Вундт, Д. Н. Узнадзе, В. Г. Леонтьев и др.) предпринимали в своих теориях попытки отразить сложность мотива как психологического образования. Все больше осознавался тот факт, что на процесс поведения и деятельности влияют не разрозненные факты, а их совокупность.
Вундт, Д. Н. Узнадзе, В. Г. Леонтьев и др.) предпринимали в своих теориях попытки отразить сложность мотива как психологического образования. Все больше осознавался тот факт, что на процесс поведения и деятельности влияют не разрозненные факты, а их совокупность.

 txtb.ru
txtb.ru
 Асах С.Т. и др.
Консерв Биол. 2013 авг; 27 (4): 866-75. doi: 10.1111/cobi.12058. Epub 2013 8 мая.
Консерв Биол. 2013.
PMID: 23656329
Асах С.Т. и др.
Консерв Биол. 2013 авг; 27 (4): 866-75. doi: 10.1111/cobi.12058. Epub 2013 8 мая.
Консерв Биол. 2013.
PMID: 23656329 2003.
PMID: 12825773
Обзор.
2003.
PMID: 12825773
Обзор. pone.0286053. Электронная коллекция 2023.
ПЛОС Один. 2023.
PMID: 37205662
Бесплатная статья ЧВК.
pone.0286053. Электронная коллекция 2023.
ПЛОС Один. 2023.
PMID: 37205662
Бесплатная статья ЧВК. 2018.
PMID: 37131403
Бесплатная статья ЧВК.
Аннотация недоступна.
2018.
PMID: 37131403
Бесплатная статья ЧВК.
Аннотация недоступна. Эта структура также участвует в регуляции продолжительности стрессовых реакций на раздражители окружающей среды.
Эта структура также участвует в регуляции продолжительности стрессовых реакций на раздражители окружающей среды.
