Генотип и фенотип. Лицо и мозг генетически связаны. Главные новости 6 апреля
Из 472 геномных регионов, ответственных за развитие мозга, 76 — ответственны и за формирование лица человека. Создана база, в которой собраны научно подтвержденные данные о том, как выглядели неандертальцы и другие гоминиды.
Владимир Губайловский
Генотип, конечно, определяет фенотип (то есть внешние признаки), но происходит это совсем не очевидным образом
Английское выражение «beauty is only skin deep» можно перевести на русский приблизительно: «красота обманчива» или «не суди по лицу». Генетики Стэнфорда решили это выражение несколько конкретизировать. Ученые рассматривали ситуацию, когда две значительно различающиеся по генотипу и фенотипу (внешним признакам) популяции людей смешиваются и скрещиваются. Сохраняется ли различие геномов этих популяций? Генетики пришли к выводу, что спустя несколько поколений геномные различия стираются. Это происходит даже в том случае, когда в качестве пары выбирается только похожий партнер.
Но с другой стороны… В Медицинской школе Стэнфорда выделили гены, ответственные за форму лица человека. Оказалось, что 76 геномных регионов, отвечающих за наше лицо, входят в состав тех 472 геномных регионов, которые отвечают за развитие и формирование мозга. Ученые не ждали такого массового пересечения. Мозг и лицо оказываются тесно связаны в своем развитии. Правда, один из авторов исследования Джоанна Высоцка говорит, что предсказывать по лицу ранний Альцгеймер или, пристально поглядев на человека, узнать его IQ, пока не получится, — за это отвечают другие гены. Но то, что лицо может многое о мозге рассказать, ученые не сомневаются.
В конечном счете внешний вид все равно формируется в основном геномом. Но процесс этого формирования крайне непрост. Резуховидка Таля или Arabidopsis thaliana — это такая мушка дрозофила для генетиков (только среди растений). У нее довольно короткий геном (150 млн пар нуклеотидов) и с ней удобно работать. Генетики Стэнфордского университета провели PHK-секвенирование (ScRNA-seq) 20 тысяч клеток на верней и нижней сторонах листа резуховидки. Ученые постарались установить, как происходит процесс дифференцировки стволовых клеток. РНК-секвенирование позволяет отследить редкие, временные изменения, возникающие при дифференцировке клеток. Оказалось, что клетки вовсе не идут строем по прямой. Они ведут себя очень индивидуально, и даже могут, как бы посомневавшись, вернуться к той форме, которую уже прошли — заложить петлю. Эта работа имеет большое значение для понимания самого процесса морфогенеза, то есть образования различных органов и тканей из эмбриональных клеток.
Но процесс этого формирования крайне непрост. Резуховидка Таля или Arabidopsis thaliana — это такая мушка дрозофила для генетиков (только среди растений). У нее довольно короткий геном (150 млн пар нуклеотидов) и с ней удобно работать. Генетики Стэнфордского университета провели PHK-секвенирование (ScRNA-seq) 20 тысяч клеток на верней и нижней сторонах листа резуховидки. Ученые постарались установить, как происходит процесс дифференцировки стволовых клеток. РНК-секвенирование позволяет отследить редкие, временные изменения, возникающие при дифференцировке клеток. Оказалось, что клетки вовсе не идут строем по прямой. Они ведут себя очень индивидуально, и даже могут, как бы посомневавшись, вернуться к той форме, которую уже прошли — заложить петлю. Эта работа имеет большое значение для понимания самого процесса морфогенеза, то есть образования различных органов и тканей из эмбриональных клеток.
Рассматривая изображения вымерших гоминид, мы составляем свое представление о них. И часто это представление не основано ни на чем, кроме воображения художника, который их нарисовал. Райн Кэмбелл антрополог из Университета Аделаиды, Австралия замечает: «Неандертальцев обычно рисуют с грязными, всклоченными волосами. Но это чистая выдумка. Мы можем сделать некоторые обоснованные предположения о цвете волос, но мы не знаем, как неандертальцы ухаживали за своими волосами и какие они использовали средства гигиены. Животные ухаживают за шерстью очень тщательно. Почему же мы отказываем в этом неандертальцам?» Антропологи из Университета Говарда в Вашингтоне и Университета Аделаиды решили поправить эту ситуацию и создали специальную базу данных, в которой собрали известные на сегодня научные представления о внешнем виде ископаемых гоминид. Ученые полагают, что правильное представление о наших предках важно и с научной, и с этической точек зрения.
И часто это представление не основано ни на чем, кроме воображения художника, который их нарисовал. Райн Кэмбелл антрополог из Университета Аделаиды, Австралия замечает: «Неандертальцев обычно рисуют с грязными, всклоченными волосами. Но это чистая выдумка. Мы можем сделать некоторые обоснованные предположения о цвете волос, но мы не знаем, как неандертальцы ухаживали за своими волосами и какие они использовали средства гигиены. Животные ухаживают за шерстью очень тщательно. Почему же мы отказываем в этом неандертальцам?» Антропологи из Университета Говарда в Вашингтоне и Университета Аделаиды решили поправить эту ситуацию и создали специальную базу данных, в которой собрали известные на сегодня научные представления о внешнем виде ископаемых гоминид. Ученые полагают, что правильное представление о наших предках важно и с научной, и с этической точек зрения.
ГЕНОТИП-ФЕНОТИПИЧЕСКИЕ КОРРЕЛЯЦИИ ТЕЧЕНИЯ КИСТОЗНОГО ФИБРОЗА У РОССИЙСКИХ ДЕТЕЙ. ПЕРВОЕ ОПИСАНИЕ ОДИННАДЦАТИ НОВЫХ МУТАЦИЙ | Горинова
1. Капранов Н.И., Каширская Н.Ю. Муковисцидоз. — М.: Медпрактика-М; 2014. — 672 с.
Капранов Н.И., Каширская Н.Ю. Муковисцидоз. — М.: Медпрактика-М; 2014. — 672 с.
2. Farrell P. The prevalence of cystic fibrosis in the European Union. J Cyst Fibros. 2008;7(5):450–453. doi: 10.1016/j.jcf.2008.03.007.
3. Красовский С.А., Черняк А.В., Каширская Н.Ю., и др. Муковисцидоз в России: Создание национального регистра // Педиатрия. Журнал им. Г.Н. Сперанского. — 2014. — Т. 93. — № 4 — С. 44–55.
4. Каширская Н.Ю., Красовский С.А., Черняк А.В., и др. Динамика продолжительности жизни больных муковисцидозом, проживающих в Москве, и ее связь с получаемой терапией: ретроспективный анализ за 1993–2013 гг. // Вопросы современной педиатрии. — 2015. — Т. 14. — № 4 — С. 503–508. doi: 10.15690/vsp.v14.i4.1390.
5. Riordan JR, Rommens JM, Kerem B, et al. Identification of the cystic fibrosis gene: cloning and characterization of complementary DNA. Science. 1989;245(4922):1066–1073. doi: 10.1126/science.2475911.
6. Kerem B, Rommens JM, Buchanan JA, et al. Identification of the cystic fibrosis gene: genetic analysis. Science. 1989; 245(4922):1073–1080. doi: 10.1126/science.2570460.
Science. 1989; 245(4922):1073–1080. doi: 10.1126/science.2570460.
7. genet.sickkids.on.ca [Internet]. Cystic Fibrosis Mutation Database [cited 2017 Dec 24]. Available from: www.genet.sickkids. on.ca/cftr.
8. Audrezet MP, Dabricot A, Le Marechal C, Ferec C. Validation of high-resolution DNA melting analysis for mutation scanning of the cystic fibrosis transmembrane conductance regulator (CFTR) gene. J Mol Diagn. 2008;10(5):424–434. doi: 10.2353/jmoldx.2008.080056.
9. Le Marechal C, Audrezet MP, Quere I, et al. Complete and rapid scanning of the cystic fibrosis transmembrane conductance regulator (CFTR) gene by denaturing high-performance liquid chromatography (DHPLC): major inplications for genetics counseling. Hum Genet. 2001;108(4):290–298. doi: 10.1007/s004390100490.
10. Galvin P, Clarke L, Harvey S, Amaral M. Microarray analysis in cystic fibrosis. J Cyst Fibros. 2004;3(2):29–33. doi: 10.1016/j.jcf.2004.05.006.
11. Баранов А.А., Капранов Н.И., Каширская Н. Ю., и др. Проблемы диагностики муковисцидоза и пути их решения в России / Педиатрическая фармакология. — 2014. — Т. 11. — № 6 — С. 16–23. doi: 10.15690/pf.v11i6.1211.
Ю., и др. Проблемы диагностики муковисцидоза и пути их решения в России / Педиатрическая фармакология. — 2014. — Т. 11. — № 6 — С. 16–23. doi: 10.15690/pf.v11i6.1211.
12. Lucarelli M, Narzi L, Piergentili R, et al. A 96-well formatted method for exon and exon/intron boundary full sequencing of the CFTR gene. Anal Biochem. 2006;353(2):226–235. doi: 10.1016/j.ab.2006.03.022.
13. A new targeted CFTR mutation panel based on next-generation sequencing technology. J Mol Diagn. 2017;19(5):788–800. doi: 10.1016/j.jmoldx.2017.06.002.
14. Schrijver I, Rappahahn K, Pique L, et al. Multiplex ligationdependent probe amplification identification of whole exon and single nucleotide deletions in the CFTR gene of Hispanic individuals with cystic fibrosis. J Mol Diagn. 2008;10(4):368–375. doi: 10.2353/jmoldx.2008.080004.
15. Mehdizadeh Hakkak A, Keramatipour M, Talebi S, et al. Analysis of CFTR gene mutations in children with cystic fibrosis, first report from North-East of Iran. Iran J Basic Med Sci.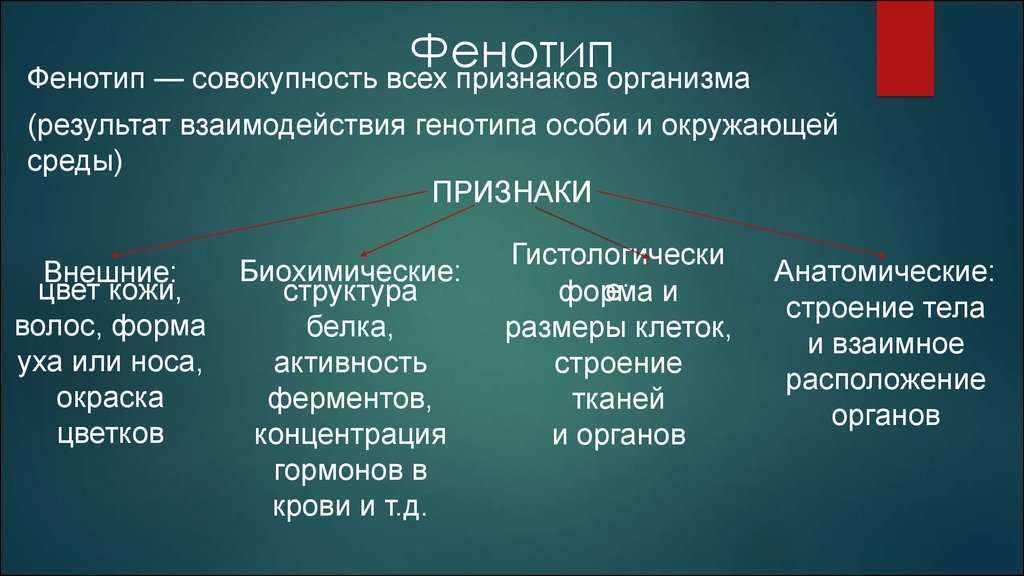 2013;16(8):918–921.
2013;16(8):918–921.
16. Регистр больных муковисцидозом в Российской Федерации. 2015 год / Под ред. Е.И. Кондратьевой, С.А. Красовского, А.Ю. Воронковой, и др. — М.: Медпрактика-М; 2016. — 72 с.
17. Gurwitz D, Corey M, Francis PW. Perspectives in cystic fibrosis. Pediatr Clin North Am. 1979;26(3):603–615. doi: 10.1016/S00313955(16)33752-X.
18. Rowntree RK, Harris A. The phenotypic consequences of CFTR mutations. Ann Hum Genet. 2003;67(Pt 5):471–485. doi: 10.1046/j.1469-1809.2003.00028.x.
19. Green DM, McDougal KE, Blackman SM, et al. Mutations that permit residual CFTR function delay acquisition of multiple respiratory pathogens in CF patients. Respir Res. 2010;11:140. doi: 10.1186/1465-9921-11-140.
20. Bombieri C, Seia M, Castellani C. Genotypes and phenotypes in cystic fibrosis and cystic fibrosis transmembrane regulator-related disorders. Semin Respir Crit Care Med. 2015;36(2):180–193. doi: 10.1055/s-0035-1547318.
21. Cutting GR. Cystic fibrosis genetics: from molecular understanding to clinical application.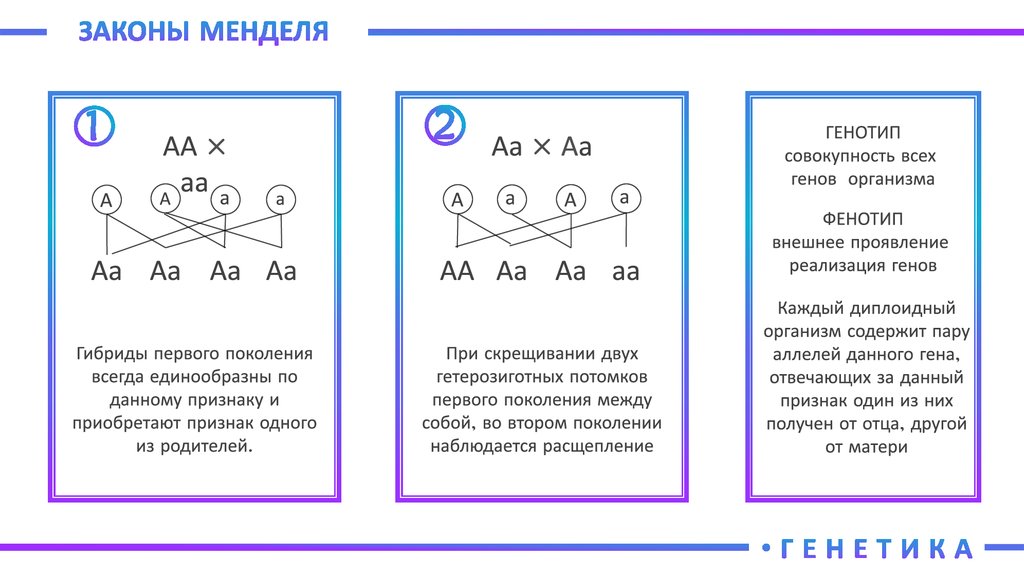 Nat Rev Genet. 2015;16(1):45–56. doi: 10.1038/nrg3849.
Nat Rev Genet. 2015;16(1):45–56. doi: 10.1038/nrg3849.
22. Elborn JS. Personalised medicine for cystic fibrosis: treating the basic defect. Eur Respir Rev. 2013;22(127):3–5. doi: 10.1183/09059180.00008112.
23. Foucher J, Chanteloup E, Vergniol J, et al. Diagnosis of cirrhosis by transient elastography (FibroScan): a prospective study. Gut. 2006;55(3):403–408. doi: 10.1136/gut.2005.069153.
24. hgmd.cf.ac.uk [Internet]. HGMD® Professional 2017.4. [cited 2018 Feb 20]. Available from: http://www.hgmd.cf.ac.uk/ac/index.php.
25. Одинокова О.Н. Расширенный поиск мутаций гена CFTR в выборке больных муковисцидозом из Сибирского региона. / VII ежегодная Северо-Западная с международным участием научно-практическая конференция по муковисцидозу «Практика лечения муковисцидоза»; Май 27–28, 2016; Санкт-Петербург. Доступно по: http://ostrovaru.com/%D0%BF%D1%80%D0%BE%D0%B3%D1%80%D0%B0%D0%BC%D0%BC%D1%8B/2015/01/08/%D0%BA%D0%BE%D0%BD%D1%84%D0%B5%D1%80%D0%B5%D0%BD%D1%86%D0%B8%D0%B8%D0%B8-%D1%81%D0%B5%D0%BC%D0%B8%D0%BD%D0%B0%D1%80%D1%8B/. Ссылка активна на 12.02.2018.
Ссылка активна на 12.02.2018.
26. Sinaasappel M, Stern M, Littlewood J, et al. Nutrition in patients with cystic fibrosis: a European Consensus. J Cyst Fibros. 2002; 1(2):51–75. doi: 10.1016/S1569-1993(02)00032-2.
27. Richards S, Aziz N, Bale S, et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 2015;17(5):405–424. doi: 10.1038/gim.2015.30.
28. Кондратьева Е.И., Шерман В.Д., Амелина Е.Л., и др. Клинико-генетическая характеристика и исходы мекониевого илеуса при муковисцидозе // Российский вестник перинатологии и педиатрии. — 2016. — Т. 61. — № 6 — С. 77–81. doi: 10.21508/1027-4065-2016-61-6-77-81.
Методы интеграции данных для выявления взаимодействий генотип-фенотип
Мецкер, М. Л. Технологии секвенирования — следующее поколение. Природа Преподобный Жене. 11 , 31–46 (2010).
КАС пабмед Google Scholar
Озсолак Ф. и Милош П. М. Секвенирование РНК: достижения, проблемы и возможности. Природа Преподобный Жене. 12 , 87–98 (2011).
КАС пабмед Google Scholar
Ван, З., Герштейн, М. и Снайдер, М. RNA-Seq: революционный инструмент для транскриптомики.
КАС пабмед Google Scholar
Лэрд, П. В. Принципы и проблемы полногеномного анализа метилирования ДНК. Природа Преподобный Жене. 11 , 191–203 (2010). Это всесторонний обзор данных анализа метилирования ДНК.
КАС пабмед Google Scholar
Park, P. J. ChIP–seq: преимущества и проблемы развивающейся технологии.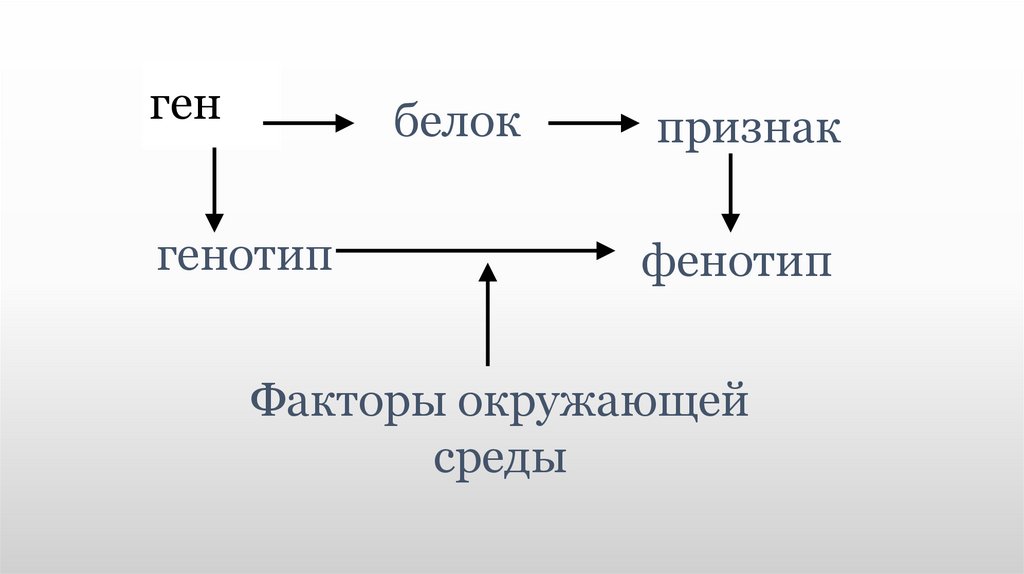 Природа Преподобный Жене. 10 , 669–680 (2009).
Природа Преподобный Жене. 10 , 669–680 (2009).
КАС пабмед Google Scholar
Алтелаар, А.Ф.М., Муньос, Дж. и Хек, А.Дж.Р. Протеомика следующего поколения: к интегративному взгляду на протеомную динамику. Природа Преподобный Жене. 14 , 35–48 (2013).
КАС пабмед Google Scholar
Шулаев В. Метаболомические технологии и биоинформатика. Краткая информация. Биоинформ. 7 , 128–139 (2006).
КАС пабмед Google Scholar
Шапиро, Э., Бизунер, Т. и Линнарссон, С. Технологии, основанные на секвенировании отдельных клеток, произведут революцию в науке о целом организме. Природа Преподобный Жене. 14 , 618–630 (2013).
КАС пабмед Google Scholar
Almasy, L.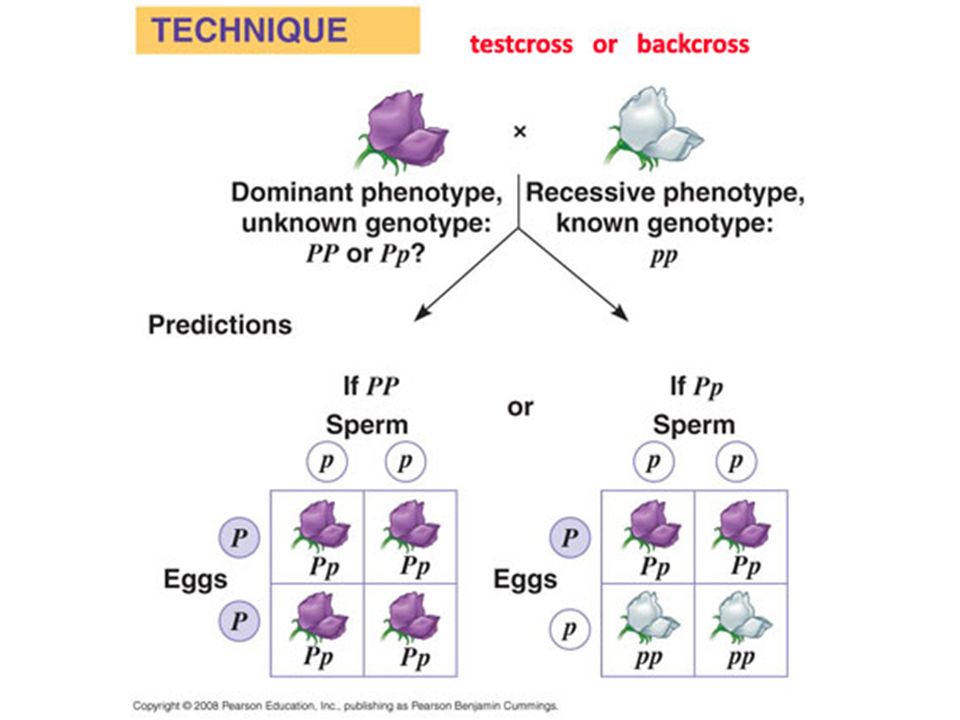 & Blangero, J. Многоточечный анализ связи количественных признаков в общих родословных. утра. Дж. Хам. Жене.
& Blangero, J. Многоточечный анализ связи количественных признаков в общих родословных. утра. Дж. Хам. Жене.
КАС пабмед ПабМед Центральный Google Scholar
Horvath, S., Xu, X. & Laird, N.M. Метод тестирования семейных ассоциаций: стратегии изучения общих генотип-фенотипических ассоциаций. Евро. Дж. Хам. Жене. 9 , 301–306 (2001).
КАС пабмед Google Scholar
Девлин, Б., Родер, К. и Бакану, С. А. Беспристрастные методы популяционных ассоциативных исследований. Жен. Эпидемиол. 21 , 273–284 (2001).
КАС пабмед Google Scholar
Рейф, Д. М., Уайт, Б. К. и Мур, Дж. Х. Комплексный анализ генетических, геномных и протеомных данных. Expert Rev. Proteomics 1 , 67–75 (2004).
КАС пабмед Google Scholar
Хамид, Дж. С. и др. Интеграция данных в генетике и геномике: методы и проблемы. Гул. Геномика Протеомика 2009 , 869093 (2009).
ПабМед ПабМед Центральный Google Scholar
Зибертс, С. К. и Шадт, Э. Э. Движение к системному генетическому взгляду на болезнь. Мамм. Геном 18 , 389–401 (2007).
ПабМед ПабМед Центральный Google Scholar
Хокинс, Р. Д., Достопочтенный, Г. К. и Рен, Б. Геномика следующего поколения: комплексный подход. Природа Преподобный Жене. 11 , 476–486 (2010).
КАС пабмед Google Scholar
Holzinger, E. R. & Ritchie, MD. Интеграция разнородных высокопроизводительных данных для метаразмерных фармакогеномных исследований и исследований, связанных с заболеваниями. Фармакогеномика 13 , 213–222 (2012).
Фармакогеномика 13 , 213–222 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Holzinger, E. et al. в Эволюционные вычисления, машинное обучение и интеллектуальный анализ данных в биоинформатике (под редакцией Джакобини, М., Ваннески, Л. и Буш, В.) 7246 , 134–143 (Springer Berlin Heidelberg, 2012).
Google Scholar
Holzinger, E. R. et al. ATHENA: инструмент метапространственного анализа, применяемый к генотипам и данным экспрессии генов для прогнозирования уровней холестерина ЛПВП. пак. Симп. Биокомпьютер. 385–396 (2013).
Штейн, Л. Д. Облачные вычисления в геномной информатике. Геном Биол. 11 , 207 (2010).
ПабМед ПабМед Центральный Google Scholar
Дорфф К. С. и др. GobyWeb: упрощенное управление и анализ экспрессии генов и данных секвенирования метилирования ДНК. PLoS ONE 8 , e69666 (2013).
С. и др. GobyWeb: упрощенное управление и анализ экспрессии генов и данных секвенирования метилирования ДНК. PLoS ONE 8 , e69666 (2013).
КАС пабмед ПабМед Центральный Google Scholar
Reid, J.G. et al. Запуск геномики в облаке: развертывание Mercury, конвейера анализа последовательностей следующего поколения. BMC Биоинформатика 15 , 30 (2014).
ПабМед ПабМед Центральный Google Scholar
Heath, A. P. et al. Bionimbus: облако для управления, анализа и обмена большими наборами геномных данных. Дж. Ам. Мед. Поставить в известность. доц. 21 , 969–975 (2014).
ПабМед ПабМед Центральный Google Scholar
Тернер, С. и др. Процедуры контроля качества полногеномных ассоциативных исследований. Курс. протокол Гум. Жене. 68 , 1.19.1–1.19.18 (2011).
Курс. протокол Гум. Жене. 68 , 1.19.1–1.19.18 (2011).
Google Scholar
Зувич Р.Л. и др. Подводные камни объединения данных GWAS: уроки, извлеченные в сети eMERGE, и процедуры контроля качества для поддержания высокого качества данных. Жен. Эпидемиол. 35 , 887–898 (2011). В этом документе представлены подробные уроки, извлеченные из процессов контроля качества высокопроизводительных данных о генотипах, и приведены рекомендации для читателей по передовым методам очистки и объединения данных о генотипах.
ПабМед ПабМед Центральный Google Scholar
Laurie, C.C. et al. Контроль качества и обеспечение качества генотипических данных для полногеномных ассоциативных исследований. Жен. Эпидемиол. 34 , 591–602 (2010).
ПабМед ПабМед Центральный Google Scholar
МакКенна, А. и др. Набор инструментов для анализа генома: платформа MapReduce для анализа данных секвенирования ДНК следующего поколения. Рез. генома. 20 , 1297–1303 (2010).
и др. Набор инструментов для анализа генома: платформа MapReduce для анализа данных секвенирования ДНК следующего поколения. Рез. генома. 20 , 1297–1303 (2010).
КАС пабмед ПабМед Центральный Google Scholar
Marguerat, S. & Bähler, J. RNA-seq: от технологии к биологии. Сотовый. Мол. Жизнь наук. 67 , 569–579 (2010).
КАС пабмед Google Scholar
Херст, М. и Марра, Массачусетс. Подходы к эпигеномике, основанные на секвенировании следующего поколения. Информационные брифинги Функц. Геном. 9 , 455–465 (2010).
КАС Google Scholar
Джонстон, И. М. и Титтерингтон, Д. М. Статистические проблемы многомерных данных. Фил. Транс. Р. Соц. А. 367 , 4237–4253 (2009).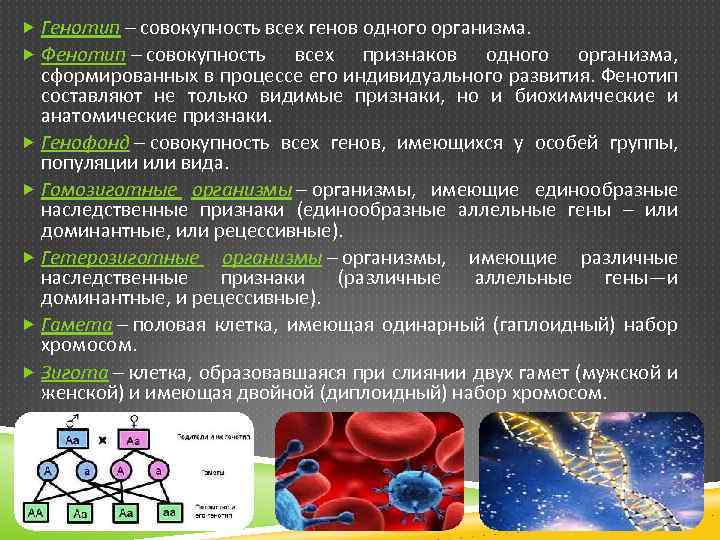
ПабМед Google Scholar
Хасти, Т., Тибширани, Р. и Фридман, Дж. Элементы статистического обучения: интеллектуальный анализ данных, вывод и прогнозирование (Springer-Verlag, 2001).
Google Scholar
Буш В. С., Дудек С. М. и Ричи М. Д. Биофильтр: система интеграции знаний для многолокусного анализа полногеномных ассоциативных исследований. пак. Симп. Биокомпьютер. 368–379 (2009).
Грин, К. С., Пенрод, Н. М., Киралис, Дж. и Мур, Дж. Х. Пространственно однородный рельеф (SURF) для эффективной в вычислительном отношении фильтрации межгенных взаимодействий. Биоданные Мин. 2 , 5 (2009).
ПабМед ПабМед Центральный Google Scholar
Мур, Дж. Х. и Уайт, Британская Колумбия, в Эволюционные вычисления, машинное обучение и интеллектуальный анализ данных в биоинформатике (редакторы Марчиори, Э. , Мур, Дж. Х. и Раджапаксе, Дж. К.) 166–175 (Springer Berlin Heidelberg, 2007).
, Мур, Дж. Х. и Раджапаксе, Дж. К.) 166–175 (Springer Berlin Heidelberg, 2007).
Google Scholar
Зоу, Х., Хасти, Т. и Тибширани, Р. Анализ разреженных главных компонентов. Дж. Вычисл. График Стат. 15 , 265–286 (2006).
Google Scholar
Холланд, Дж. Х. Генетические алгоритмы. Науч. Являюсь. 267 , 66–72 (1992).
Google Scholar
Вильхьялмссон, Б. Дж. и Нордборг, М. Природа смешения в полногеномных ассоциативных исследованиях. Природа Преподобный Жене. 14 , 1–2 (2013).
ПабМед Google Scholar
Zhou, X. & Stephens, M. Эффективные алгоритмы многомерных линейных смешанных моделей для полногеномных ассоциативных исследований. Nature Methods 11 , 407–409 (2014).
Nature Methods 11 , 407–409 (2014).
КАС пабмед ПабМед Центральный Google Scholar
Прайс, А. Л. и др. Анализ основных компонентов корректирует стратификацию в полногеномных ассоциативных исследованиях. Природа Жене. 38 , 904–909 (2006).
КАС пабмед Google Scholar
Лик, Дж. Т. и Стори, Дж. Д. Выявление гетерогенности в исследованиях экспрессии генов с помощью анализа суррогатных переменных. Генетика PLoS. 3 , e161 (2007).
Центральный пабмед Google Scholar
Hartford, C.M. et al. Популяционно-специфические генетические варианты, важные для восприимчивости к цитотоксичности цитарабинарабинозида. Кровь 113 , 2145–2153 (2009).
КАС пабмед ПабМед Центральный Google Scholar
Huang, R.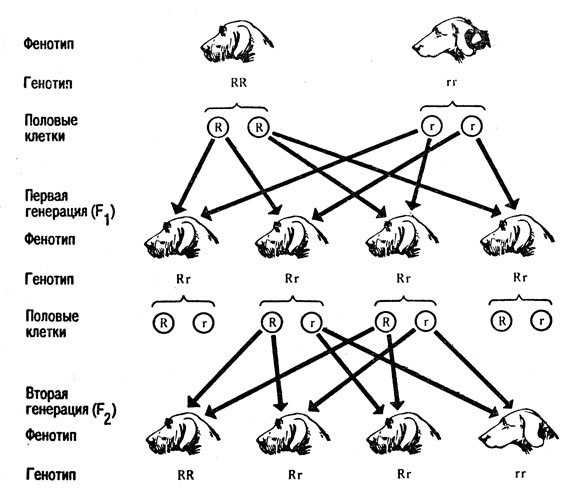 S. et al. Полногеномный подход к выявлению генетических вариантов, которые способствуют индуцированной этопозидом цитотоксичности. Проц. Натл акад. науч. США 104 , 9758–9763 (2007 г.). Это одна из первых статей, в которой представлен интегративный анализ для выявления вариантов ДНК и экспрессии генов, связанных с цитотоксичностью, вызванной химиотерапевтическими препаратами.
S. et al. Полногеномный подход к выявлению генетических вариантов, которые способствуют индуцированной этопозидом цитотоксичности. Проц. Натл акад. науч. США 104 , 9758–9763 (2007 г.). Это одна из первых статей, в которой представлен интегративный анализ для выявления вариантов ДНК и экспрессии генов, связанных с цитотоксичностью, вызванной химиотерапевтическими препаратами.
КАС пабмед Google Scholar
Huang, R. S., Duan, S., Kistner, E. O., Hartford, C. M. & Dolan, M. E. Генетические варианты, связанные с индуцированной карбоплатином цитотоксичностью в клеточных линиях, полученных от африканцев. Мол. Рак Тер. 7 , 3038–3046 (2008 г.).
КАС пабмед ПабМед Центральный Google Scholar
Schadt, E.E. et al. Интегративный геномный подход к выводу о причинно-следственных связях между экспрессией генов и заболеванием.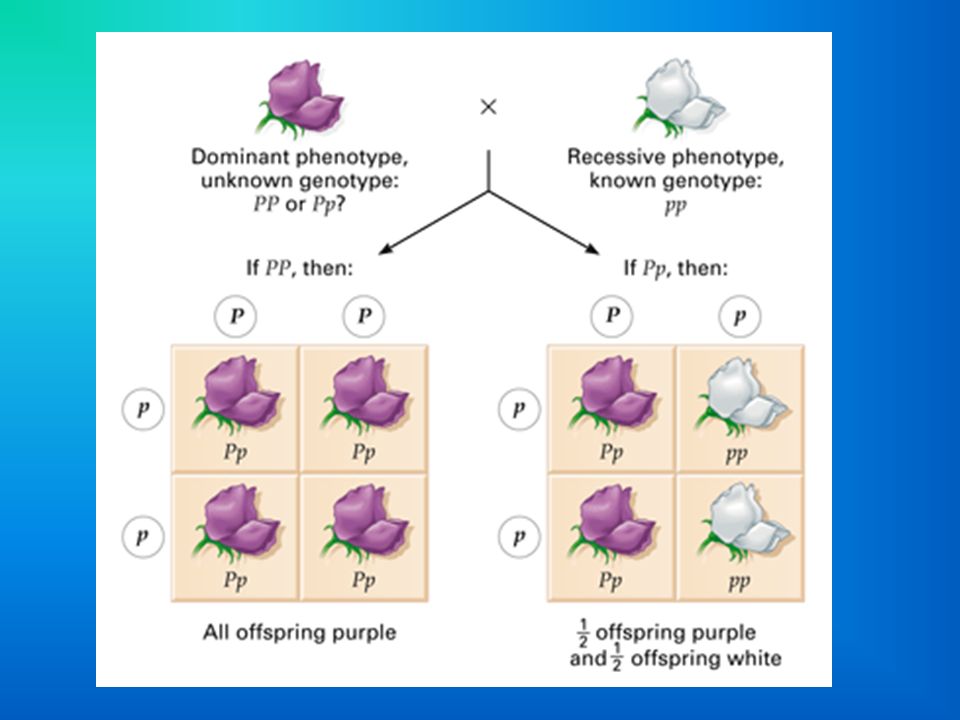 Природа Жене. 37 , 710–717 (2005). В этом исследовании использовался интегративный подход к использованию данных об вариациях ДНК и экспрессии генов для выявления драйверов сложных признаков.
Природа Жене. 37 , 710–717 (2005). В этом исследовании использовался интегративный подход к использованию данных об вариациях ДНК и экспрессии генов для выявления драйверов сложных признаков.
КАС пабмед Google Scholar
Лю, Ю. и др. Данные эпигеномной ассоциации предполагают, что метилирование ДНК является посредником генетического риска при ревматоидном артрите. Природные биотехнологии. 31 , 142–147 (2013).
КАС Google Scholar
Хан, З. и др. Количественное измерение экспрессии аллель-специфического белка в диплоидном гибриде дрожжей методом ЖХ-МС. Мол. Сист. биол. 8 , 602 (2012).
ПабМед ПабМед Центральный Google Scholar
Wei, X. & Wang, X. Вычислительный рабочий процесс для определения аллель-специфической экспрессии и эпигенетической модификации кукурузы.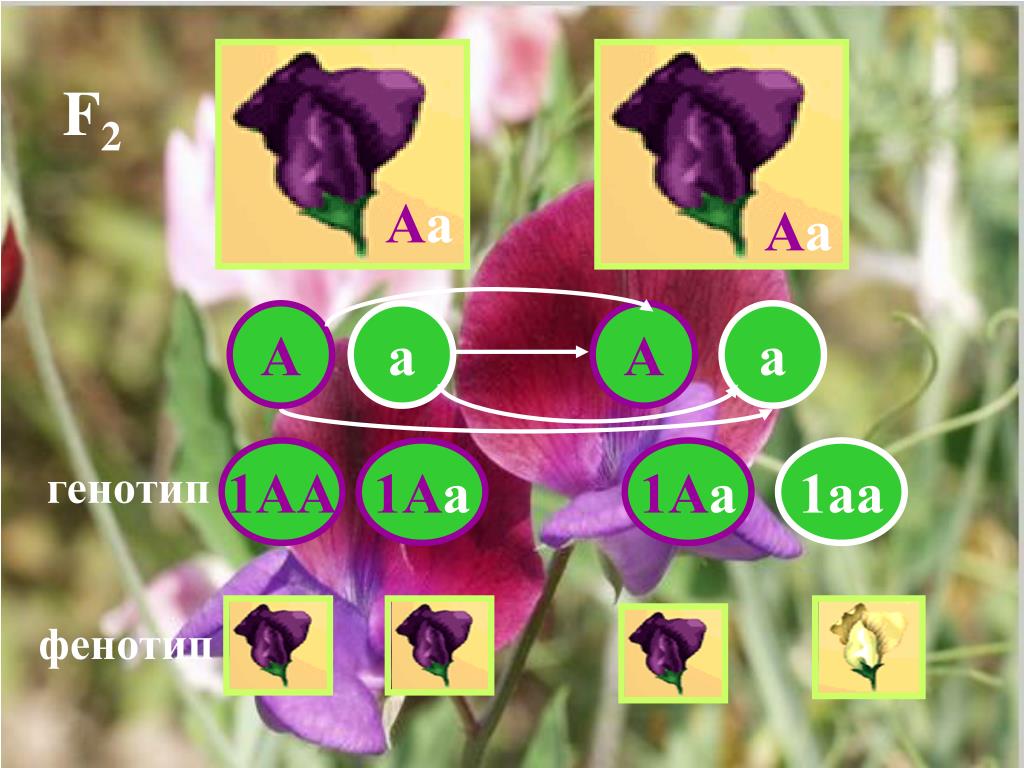 Геномика Протеомика Биоинформатика 11 , 247–252 (2013).
Геномика Протеомика Биоинформатика 11 , 247–252 (2013).
ПабМед ПабМед Центральный Google Scholar
Лаппалайнен, Т. и др. Секвенирование транскриптома и генома раскрывает функциональные различия у людей. Природа 501 , 506–511 (2013). В этом документе сообщается о секвенировании и анализе мРНК и микроРНК сотен представителей различных этнических групп в рамках проекта «1000 геномов».
КАС пабмед ПабМед Центральный Google Scholar
Мейнард, Н.Д., Чен, Дж., Стюарт, Р.К., Фан, Дж.-Б. и Рен, Б. Полногеномное картирование аллель-специфических взаимодействий белок-ДНК в клетках человека. Nature Methods 5 , 307–309 (2008).
КАС пабмед Google Scholar
Kasowski, M. et al. Обширные различия в состояниях хроматина у людей. Наука 342 , 750–752 (2013).
et al. Обширные различия в состояниях хроматина у людей. Наука 342 , 750–752 (2013).
КАС пабмед ПабМед Центральный Google Scholar
McVicker, G. et al. Идентификация генетических вариантов, влияющих на модификации гистонов в клетках человека. Наука 342 , 747–749 (2013).
КАС пабмед ПабМед Центральный Google Scholar
Консорциум проектов Encode. Проект ENCODE (ENCyclopedia Of DNA Elements). Наука 306 , 636–640 (2004).
Канехиса, М. и Гото, С. КЭГГ: Киотская энциклопедия генов и геномов. Рез. нуклеиновых кислот. 28 , 27–30 (2000).
КАС пабмед ПабМед Центральный Google Scholar
Ким Д., Шин Х., Сонг Ю. С.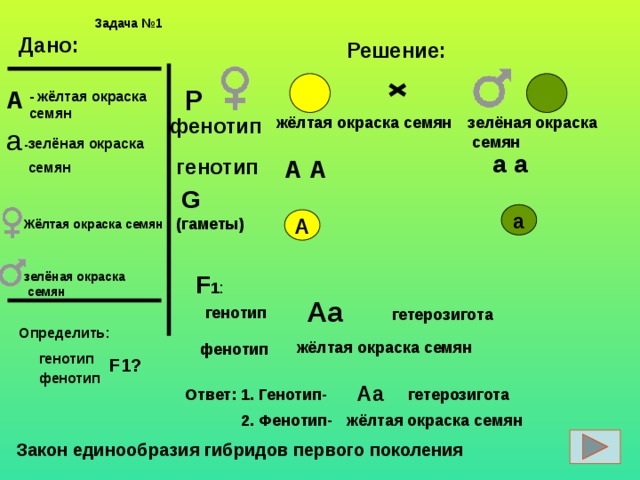 и Ким Дж. Х. Синергетический эффект различных уровней геномных данных для прогнозирования клинических исходов рака. Дж. Биомед. Поставить в известность. 45 , 1191–1198 (2012). В этом исследовании показан основанный на графах подход к прогнозированию клинического исхода рака путем интеграции мультиомных данных в виде интеграции на основе преобразования.
и Ким Дж. Х. Синергетический эффект различных уровней геномных данных для прогнозирования клинических исходов рака. Дж. Биомед. Поставить в известность. 45 , 1191–1198 (2012). В этом исследовании показан основанный на графах подход к прогнозированию клинического исхода рака путем интеграции мультиомных данных в виде интеграции на основе преобразования.
КАС пабмед Google Scholar
Фридли Б.Л., Лунд С., Дженкинс Г.Д. и Ван Л.А. Байесовская интегративная геномная модель для анализа путей сложных признаков. Жен. Эпидемиол. 36 , 352–359 (2012).
ПабМед ПабМед Центральный Google Scholar
Mankoo, P.K., Shen, R., Schultz, N., Levine, D.A. & Sander, C. Время до рецидива и выживаемость при серозных опухолях яичников, предсказанные на основе интегрированных геномных профилей. PLoS ONE 6 , e24709 (2011).
PLoS ONE 6 , e24709 (2011).
КАС пабмед ПабМед Центральный Google Scholar
Хользингер, Э. Р., Дудек, С. М., Фрейз, А. Т., Пендерграсс, С. А. и Ричи, М. Д. ATHENA: инструмент анализа наследственных и экологических сетевых ассоциаций. Биоинформатика 30 , 698–705 (2014). ATHENA — это инструмент для метапространственной интеграции мультиомных данных. В этой статье описывается программное обеспечение и его применение для этих типов анализа.
КАС пабмед Google Scholar
Ким, Д., Ли, Р., Дудек, С. М. и Ричи, доктор медицинских наук ATHENA: Выявление взаимодействий между различными уровнями геномных данных, связанных с клиническими исходами рака, с использованием нейронной сети грамматической эволюции. Биоданные Мин. 6 , 23 (2013).
ПабМед ПабМед Центральный Google Scholar
Clarke, R. et al. Свойства многомерных пространств данных: последствия для изучения данных об экспрессии генов и белков. Nature Rev. Рак 8 , 37–49 (2008). В этом обзоре рассматриваются свойства многомерных пространств данных и проблемы анализа и интерпретации данных.
et al. Свойства многомерных пространств данных: последствия для изучения данных об экспрессии генов и белков. Nature Rev. Рак 8 , 37–49 (2008). В этом обзоре рассматриваются свойства многомерных пространств данных и проблемы анализа и интерпретации данных.
КАС Google Scholar
Ланкриет, Г. Р. Г., Де Би, Т., Кристианини, Н., Джордан, М. И. и Ноубл, В. С. Статистическая основа для слияния геномных данных. Биоинформатика 20 , 2626–2635 (2004). Это первое исследование, предлагающее интеграцию на основе ядра как интеграцию на основе преобразования.
КАС пабмед Google Scholar
Боргвардт, К. М. и др. Прогнозирование функции белка с помощью ядер графа. Биоинформатика 21 , i47–i56 (2005).
КАС пабмед Google Scholar
Цуда, К. , Шин, Х. и Шёлкопф, Б. Классификация быстрых белков с несколькими сетями. Биоинформатика 21 , ii59–ii65 (2005).
, Шин, Х. и Шёлкопф, Б. Классификация быстрых белков с несколькими сетями. Биоинформатика 21 , ii59–ii65 (2005).
КАС пабмед Google Scholar
Шин Х., Лисевски А. М. и Лихтарге О. Повышение резкости графиков плюс интеграция графиков: синергия, улучшающая функциональную классификацию белков. Биоинформатика 23 , 3217–3224 (2007).
КАС пабмед Google Scholar
Turner, S.D., Dudek, S.M. & Ritchie, MD. ATHENA: основанный на знаниях гибридный алгоритм обратного распространения и грамматической эволюции для обнаружения эпистаза среди локусов количественных признаков. Мин. биоданных 3 , 5 (2010).
ПабМед ПабМед Центральный Google Scholar
Драгичи С. и Поттер Р. Б. Прогнозирование лекарственной устойчивости ВИЧ с помощью нейронных сетей. Биоинформатика 19 , 98–107 (2003).
Биоинформатика 19 , 98–107 (2003).
Google Scholar
Шен, Х.-Б. и Чоу, К.-К. Ансамбльный классификатор для распознавания образов белковых складок. Биоинформатика 22 , 1717–1722 (2006).
КАС пабмед Google Scholar
Akavia, U.D. et al. Комплексный подход к выявлению факторов, вызывающих рак. Cell 143 , 1005–1017 (2010). В этой статье продемонстрирована вычислительная структура, которая идентифицирует драйверы меланомы с использованием числа копий хромосом и данных об экспрессии генов.
КАС пабмед ПабМед Центральный Google Scholar
Zhu, J. et al. Сшивание вместе нескольких измерений данных выявляет взаимодействующие метаболомные и транскриптомные сети, которые модулируют клеточную регуляцию.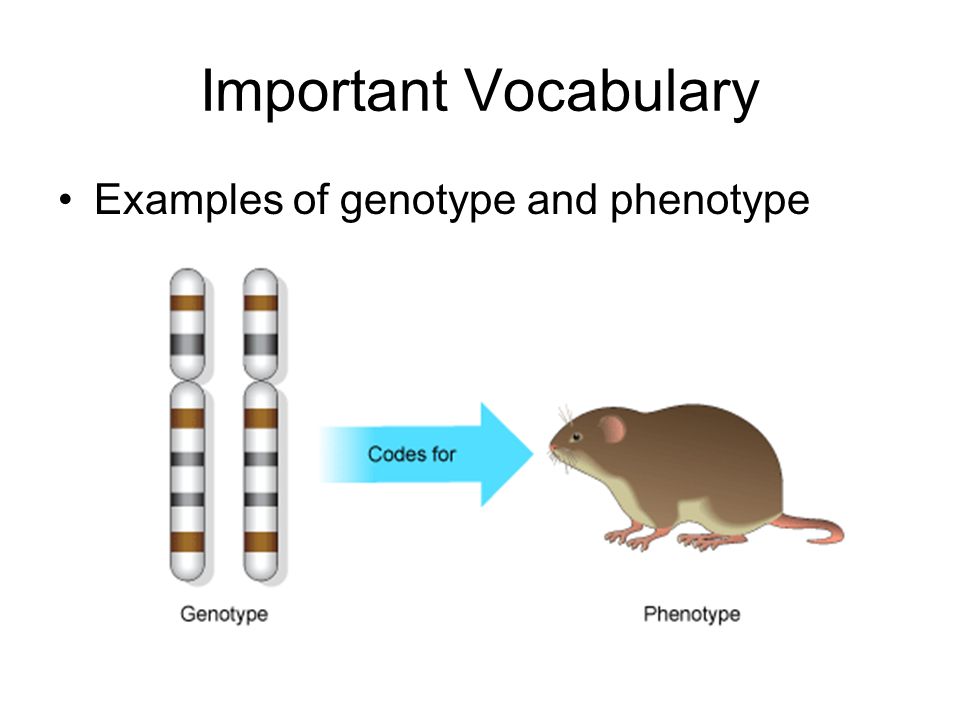 PLoS Биол. 10 , e1001301 (2012).
PLoS Биол. 10 , e1001301 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Zhu, J. et al. Интеграция крупномасштабных функциональных геномных данных для анализа сложности регуляторных сетей дрожжей. Природа Жене. 40 , 854–861 (2008).
КАС пабмед Google Scholar
Опиц Д. и Маклин Р. Популярные ансамблевые методы: эмпирическое исследование. Дж. Артиф. Интел. Рез. 11 , 169–198 (1999).
Google Scholar
Shen, R. et al. Интегративное обнаружение подтипа глиобластомы с использованием iCluster. ПЛОС ОДИН 7 , e35236 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Кирк П.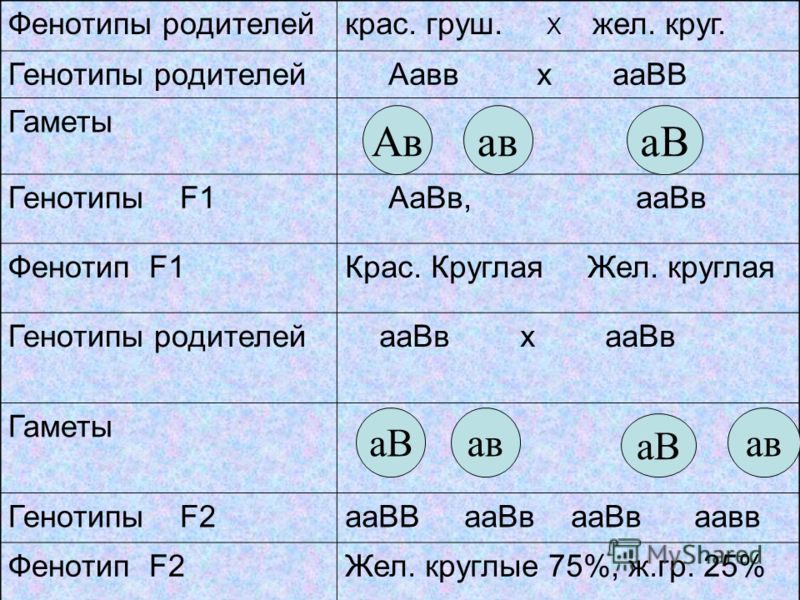 , Гриффин Дж. Э., Сэвидж Р. С., Гахрамани З. и Уайлд Д. Л. Байесовская коррелированная кластеризация для интеграции нескольких наборов данных. Биоинформатика 28 , 3290–3297 (2012).
, Гриффин Дж. Э., Сэвидж Р. С., Гахрамани З. и Уайлд Д. Л. Байесовская коррелированная кластеризация для интеграции нескольких наборов данных. Биоинформатика 28 , 3290–3297 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Лок, Э. Ф. и Дансон, Д. Б. Байесовская консенсусная кластеризация. Биоинформатика 29 , 2610–2616 (2013).
КАС пабмед ПабМед Центральный Google Scholar
Dupont, W.D. & Plummer, W.D. Расчет мощности и размера выборки. Обзор и компьютерная программа. Контрольная клин. Испытания 11 , 116–128 (1990).
КАС пабмед Google Scholar
Рабочая группа NCI–NHGRI по репликации в ассоциативных исследованиях. Воспроизведение ассоциаций генотип-фенотип. Природа 447 , 655–660 (2007).
Природа 447 , 655–660 (2007).
Грин, К. С., Пенрод, Н. М., Уильямс, С. М. и Мур, Дж. Х. Неспособность воспроизвести генетическую ассоциацию может дать важные сведения о генетической архитектуре. PLoS ONE 4 , e5639 (2009).
ПабМед ПабМед Центральный Google Scholar
Ciesielski, T. et al. Разнообразные конвергентные данные в генетическом анализе сложных заболеваний: координация омических, информационных и экспериментальных данных для лучшего выявления и проверки факторов риска. Мин. биоданных 7 , 10 (2014).
ПабМед ПабМед Центральный Google Scholar
Van Poucke, M., Vanhaesebrouck, A.E., Peelman, L.J. & Van Ham, L. Экспериментальная проверка в Silico Предсказано KCNA1 , KCNA2 , KCNA6 и KCNQ2 5 , KCNA6 и KCNQ2 5 , KCNA6 и KCNQ2 5 5.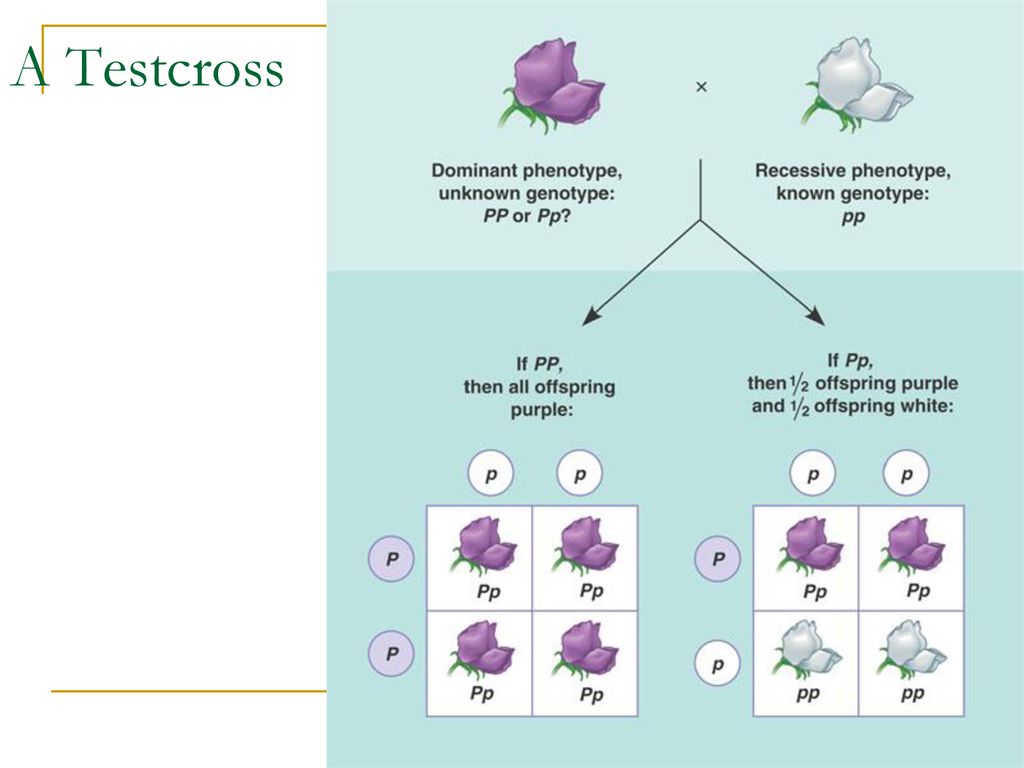 Синдром повышенной возбудимости периферических нервов у джек-рассел-терьеров. Нервно-мышечная. Беспорядок. 22 , 558–565 (2012).
Синдром повышенной возбудимости периферических нервов у джек-рассел-терьеров. Нервно-мышечная. Беспорядок. 22 , 558–565 (2012).
ПабМед Google Scholar
Шараф, Р. Н. и др. Вычислительное предсказание и экспериментальная проверка связи FABP-1 и аденокарциномы поджелудочной железы с диабетом. ВМС Гастроэнтерол. 11 , 5 (2011).
ПабМед ПабМед Центральный Google Scholar
Raychaudhuri, S. et al. Выявление взаимосвязей между участками геномных заболеваний: прогнозирование генов в патогенных ассоциациях SNP и редких делеций. Генетика PLoS. 5 , e1000534 (2009 г.).
ПабМед ПабМед Центральный Google Scholar
Crooke, P. S. et al. Эстрогены, варианты ферментов и рак молочной железы: модель риска. Рак Эпидемиол. Биомаркеры Пред. 15 , 1620–1629 (2006).
Рак Эпидемиол. Биомаркеры Пред. 15 , 1620–1629 (2006).
КАС пабмед Google Scholar
Фаррар, Д. Э. и Глаубер, Р. Р. Мультиколлинеарность в регрессионном анализе: новый взгляд на проблему. Ред. Экон. Стат. 49 , 92 (1967).
Google Scholar
Фосетт Т. Введение в ROC-анализ. Распознавание образов. лат. 27 , 861–874 (2006).
Google Scholar
Мур, Дж. Х., Хилл, Д. П., Суловари, А. и Кидд, Л. К. в Genetic Programming Theory and Practice X 87–101 (Springer, 2013).
Google Scholar
Джин, Ю. и Сендхофф, Б. Многоцелевое машинное обучение на основе Парето: обзор тематических исследований. IEEE Trans. Сист.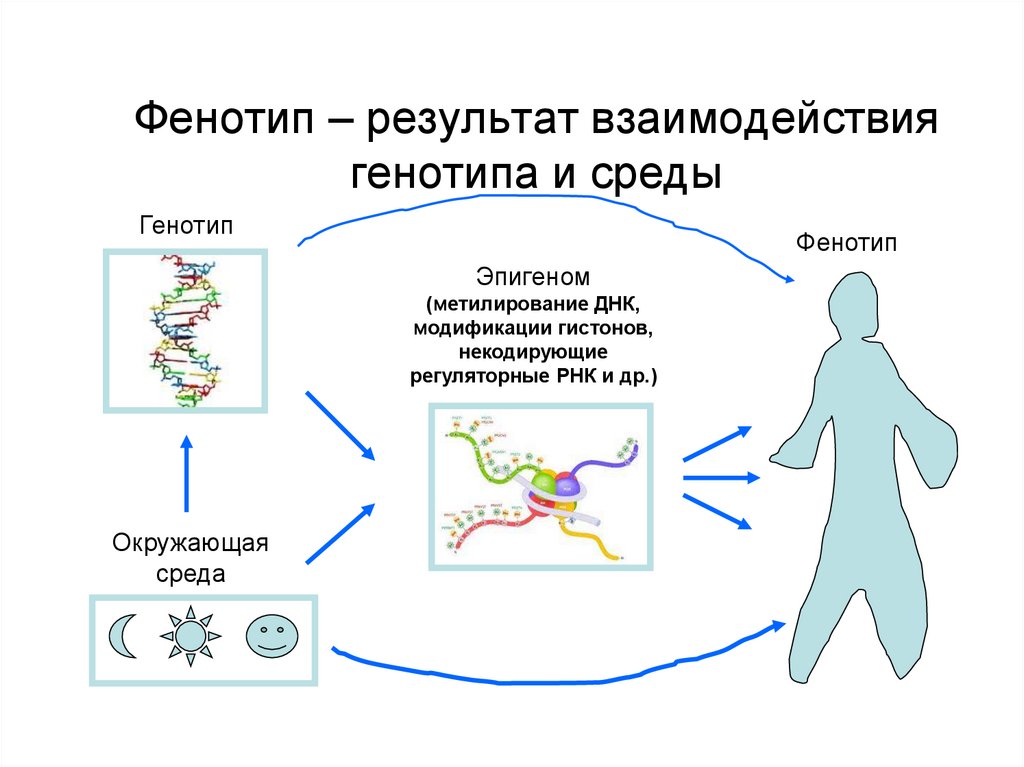 Человек Киберн. Приложение C Ред. 38 , 397–415 (2008).
Человек Киберн. Приложение C Ред. 38 , 397–415 (2008).
Google Scholar
Кристенсен В. Н. и Борресен-Дейл А. Л. Молекулярная эпидемиология рака молочной железы: генетическая изменчивость метаболизма стероидных гормонов. Мутат. Рез. 462 , 323–333 (2000).
КАС пабмед Google Scholar
Митрунен, К. и др. Глутатион S -трансфераза M1, M3, P1 и T1 генетический полиморфизм и предрасположенность к раку молочной железы. Рак Эпидемиол. Биомаркеры Пред. 10 , 229–236 (2001).
КАС пабмед Google Scholar
Киётани, К. и др. Полногеномное ассоциативное исследование идентифицирует локус 10q22, связанный с клиническими результатами адъювантной терапии тамоксифеном у пациентов с раком молочной железы в Японии. Гул. Мол. Жене. 21 , 1665–1672 (2012).
Гул. Мол. Жене. 21 , 1665–1672 (2012).
КАС пабмед Google Scholar
Garcia-Closas, M. et al. Полногеномные ассоциативные исследования идентифицируют четыре ER-отрицательных специфических локуса риска рака молочной железы. Природа Жене. 45 , 392–398, 398e1–2 (2013).
КАС пабмед Google Scholar
Михайлиду, К. и др. Крупномасштабное генотипирование выявило 41 новый локус, связанный с риском развития рака молочной железы. Природа Жене. 45 , 353–361, 361e1–2 (2013).
КАС пабмед Google Scholar
Zheng, W. et al. Общие генетические детерминанты риска рака молочной железы у женщин Восточной Азии: совместное исследование 23 637 случаев рака молочной железы и 25 579контролирует.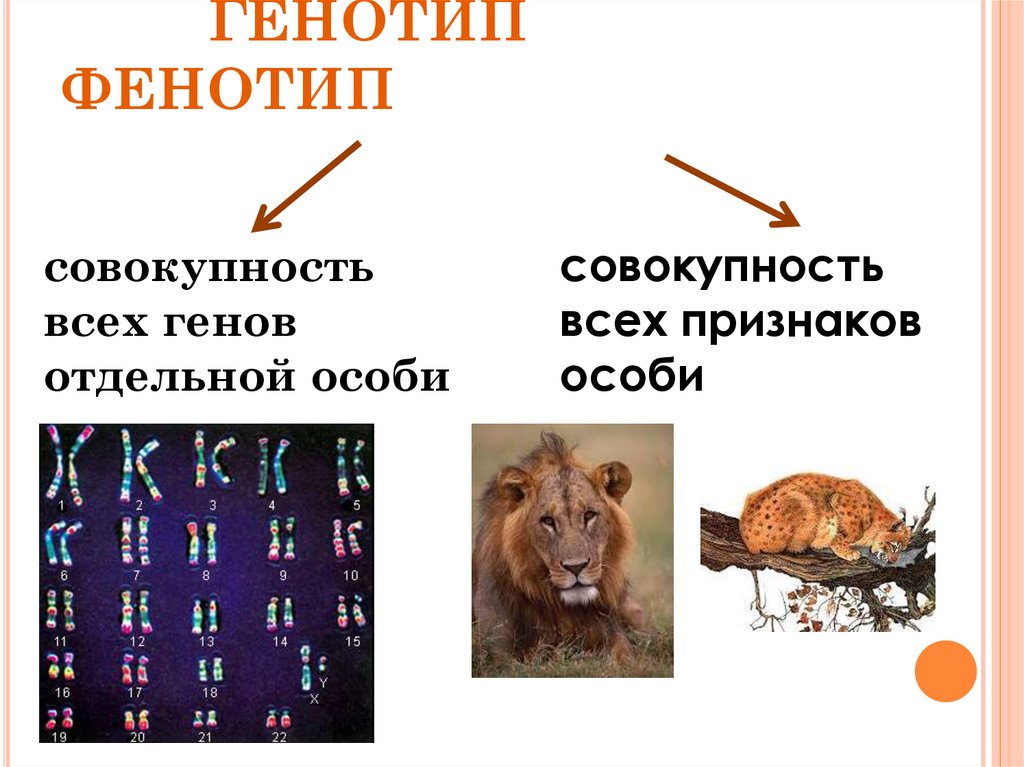 Гул. Мол. Жене. 22 , 2539–2550 (2013).
Гул. Мол. Жене. 22 , 2539–2550 (2013).
КАС пабмед ПабМед Центральный Google Scholar
Могуши, К. и Танака, Х. PathAct: новый метод анализа путей с использованием профилей экспрессии генов. Биоинформация 9 , 394–400 (2013).
ПабМед ПабМед Центральный Google Scholar
Чанг, Р.-Х. и Чен, Ю.-Э. Двухэтапный метод анализа пути на основе случайного леса. PLoS ONE 7 , e36662 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Бейли, Л. Р., Руди, Н., Дюпон, В. Д. и Парл, Ф. Ф. Связь полиморфизма цитохрома P450 1B1 (CYP1B1) со статусом стероидных рецепторов при раке молочной железы. Рак Рез. 58 , 5038–5041 (1998).
КАС пабмед Google Scholar
Шабалин А.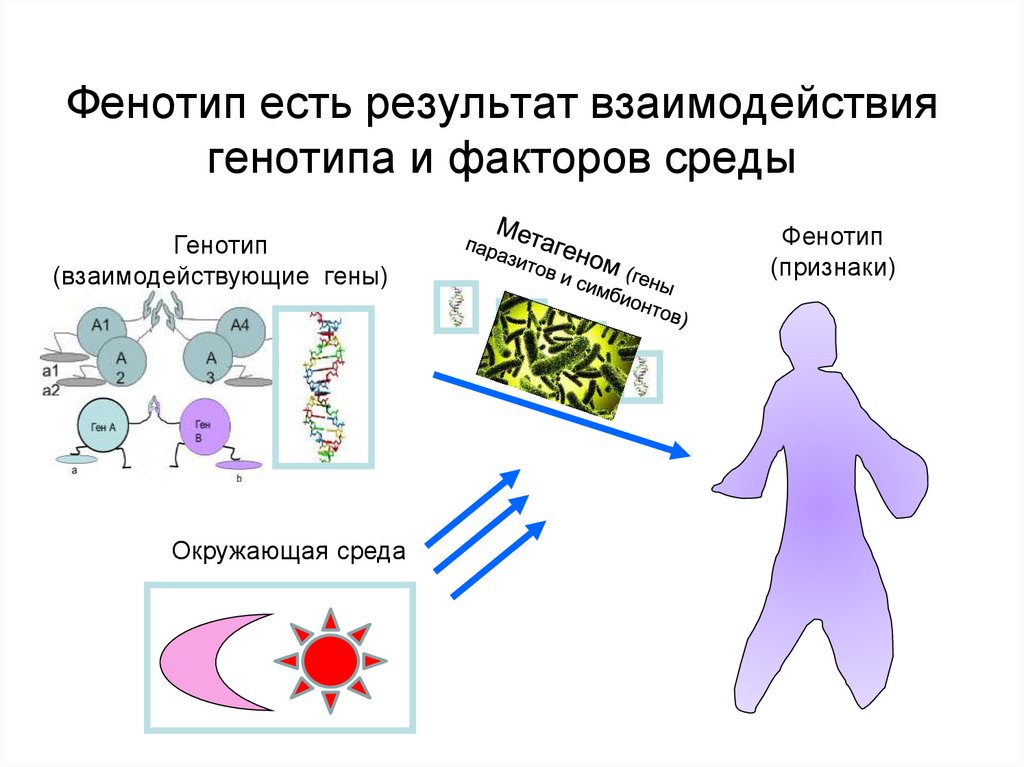 А. Matrix eQTL: сверхбыстрый анализ eQTL с помощью больших матричных операций. Биоинформатика 28 , 1353–1358 (2012).
А. Matrix eQTL: сверхбыстрый анализ eQTL с помощью больших матричных операций. Биоинформатика 28 , 1353–1358 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Абеказис Г. Р., Кардон Л. Р. и Куксон В. О. Общий тест ассоциации количественных признаков в нуклеарных семьях. утра. Дж. Хам. Жене. 66 , 279–292 (2000).
КАС пабмед Google Scholar
Розовски Дж. и др. AlleleSeq: анализ аллель-специфической экспрессии и связывания в сетевой структуре. Мол. Сист. биол. 7 , 522 (2011).
ПабМед ПабМед Центральный Google Scholar
Ван, К., Ли, М. и Хаконарсон, Х. ANNOVAR: функциональная аннотация генетических вариантов на основе данных высокопроизводительного секвенирования. Рез. нуклеиновых кислот. 38 , e164 (2010).
Рез. нуклеиновых кислот. 38 , e164 (2010).
ПабМед ПабМед Центральный Google Scholar
Ward, L. D. & Kellis, M. HaploReg: ресурс для изучения состояний хроматина, сохранения и изменений регуляторных мотивов в наборах генетически связанных вариантов. Рез. нуклеиновых кислот. 40 , Д930–Д934 (2012 г.).
КАС пабмед Google Scholar
Boyle, A. P. et al. Аннотация функциональных вариаций в личных геномах с использованием RegulomeDB. Рез. генома. 22 , 1790–1797 (2012).
КАС пабмед ПабМед Центральный Google Scholar
Эмильссон, В. и др. Генетика экспрессии генов и ее влияние на болезни. Природа 452 , 423–428 (2008). В этом важном документе представлена взаимосвязь между генетической изменчивостью, экспрессией генов и клиническими фенотипами с использованием человеческой крови и жировой ткани.
КАС пабмед Google Scholar
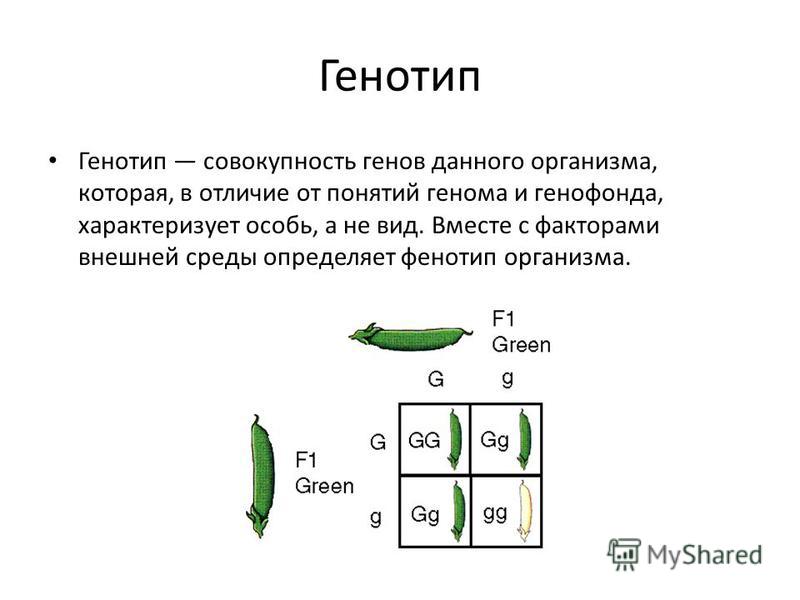
Аллели, генотип и фенотип | Science Primer
Генетика — это наука об организации, выражении и передаче наследуемой информации. Способность информации передаваться от поколения к поколению требует механизма. Живые организмы используют ДНК. ДНК представляет собой цепь или полимер нуклеиновых кислот. Отдельные полимеры ДНК могут содержать сотни миллионов молекул нуклеиновых кислот. Эти длинные нити ДНК называются хромосомами. Порядок отдельных нуклеиновых кислот в цепи содержит информацию, используемую организмами для роста и размножения. Использование ДНК в качестве информационной молекулы является универсальным свойством всего живого на Земле. Наш клеточный механизм считывает эту генетическую информацию, позволяя нашему телу синтезировать множество ферментов и белков, необходимых для жизни
На иллюстрации исследуется взаимосвязь между наличием различных аллелей в определенном локусе и генотипом и фенотипом организма.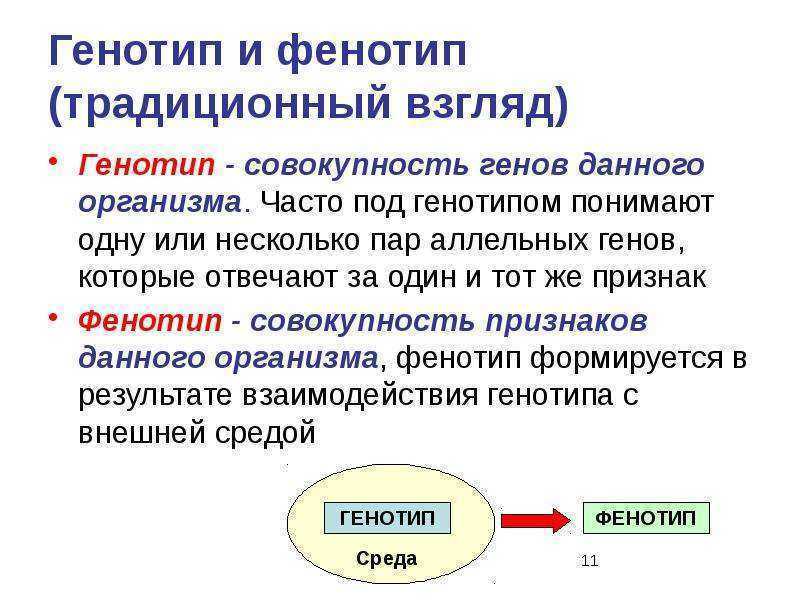 Организм в модели – растение. Он диплоидный, и признак — цвет цветка. Ниже приведено видео на YouTube, демонстрирующее использование иллюстрации и набора задач, которые вы можете использовать, чтобы проверить свое понимание этих концепций.
Организм в модели – растение. Он диплоидный, и признак — цвет цветка. Ниже приведено видео на YouTube, демонстрирующее использование иллюстрации и набора задач, которые вы можете использовать, чтобы проверить свое понимание этих концепций.
Генетическая информация хранится в дискретных единицах, называемых генами. Каждый ген содержит информацию, необходимую для синтеза отдельных клеточных компонентов, необходимых для выживания. Скоординированная экспрессия множества различных генов отвечает за рост и активность организма.
В пределах отдельного вида гены встречаются в определенных местах на хромосомах. Это позволяет определить их местонахождение. Положение определенного гена на хромосоме называется его локусом.
Вариации порядка расположения нуклеиновых кислот в молекуле ДНК позволяют генам кодировать достаточно информации для синтеза огромного разнообразия различных белков и ферментов, необходимых для жизни. Помимо различий между генами, расположение нуклеиновых кислот может различаться между копиями одного и того же гена. Это приводит к различным формам отдельных генов. Различные формы гена называются аллелями.
Это приводит к различным формам отдельных генов. Различные формы гена называются аллелями.
Организмы, размножающиеся половым путем, получают по одной полной копии своего генетического материала от каждого родителя. Наличие двух полных копий их генетического материала делает их диплоидными. Совпадающие хромосомы от каждого родителя называются гомологичными хромосомами. Совпадающие гены от каждого родителя встречаются в одном и том же месте на гомологичных хромосомах.
Диплоидный организм может иметь либо две копии одного и того же аллеля, либо по одной копии каждого из двух разных аллелей. Люди, у которых есть две копии одного и того же аллеля, считаются гомозиготными по этому локусу. Люди, которые получают разные аллели от каждого родителя, считаются гетерозиготными по этому локусу. Аллели, которые человек имеет в локусе, называются генотипом. Генотип организма часто выражается буквами. Видимое выражение генотипа называется фенотипом организма.
Аллели не созданы равными.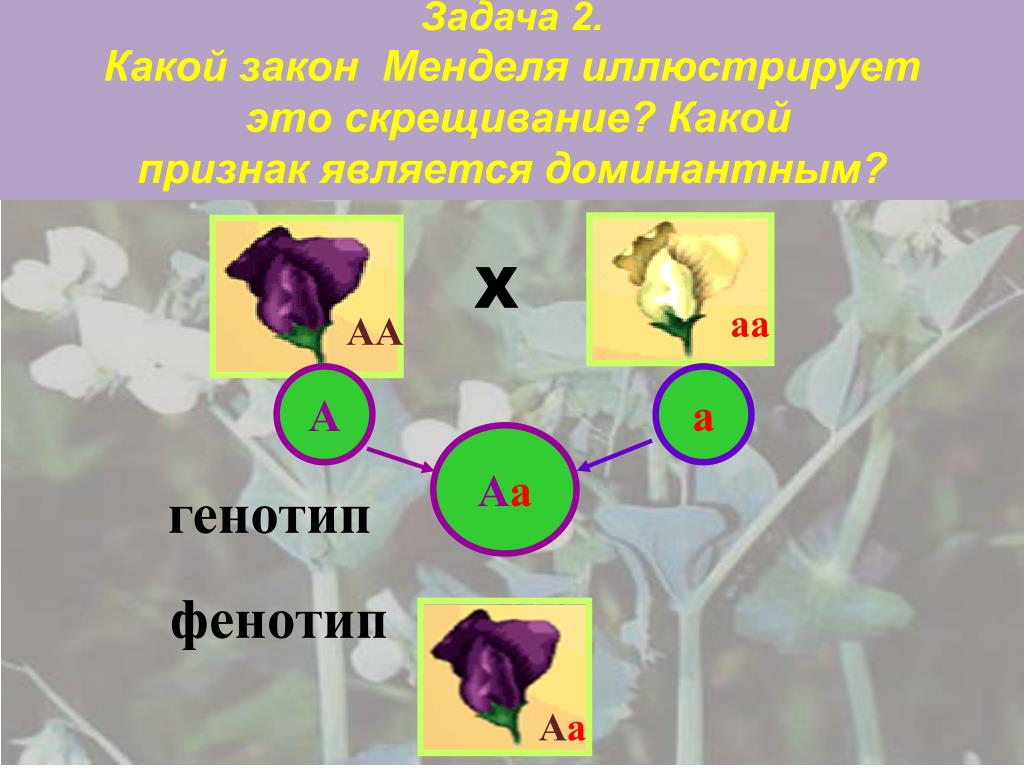 Одни аллели маскируют присутствие других. Аллели, которые маскируются другими, называются рецессивными аллелями. Рецессивные аллели экспрессируются только тогда, когда организм гомозиготен по этому локусу. Аллели, экспрессирующиеся независимо от присутствия других аллелей, называются доминантными.
Одни аллели маскируют присутствие других. Аллели, которые маскируются другими, называются рецессивными аллелями. Рецессивные аллели экспрессируются только тогда, когда организм гомозиготен по этому локусу. Аллели, экспрессирующиеся независимо от присутствия других аллелей, называются доминантными.
Если один аллель полностью маскирует присутствие другого в том же локусе, говорят, что этот аллель проявляет полное доминирование. Однако доминирование не всегда является полным. В случаях неполного доминирования возможны промежуточные фенотипы.
Взаимодействия генов могут быть довольно сложными. Приведенный выше пример демонстрирует простую ситуацию, в которой отдельному признаку соответствует один ген. В более сложных случаях несколько генов могут влиять на отдельный признак. Это называется полигенным наследованием. В этих ситуациях связь между конкретными аллелями и характеристиками не так однозначна.
В своих знаменитых исследованиях растений гороха Мендель изучил семь признаков, обладающих характеристиками, необходимыми для наблюдения за наследованием отдельных признаков.